|
生醫人的AI系列課程

課程主題一:AI與數據分析的基本概念 講師: 長庚大學資工系 李季青副教授 課程簡介: 數據分析是一門通過數學、統計學和計算機技術來解析數據的學科。它的核心在於從大量的數據中提取有價值的信息,幫助我們做出更明智的決策。數據分析的過程通常包括以下幾個步驟:數據收集、數據清理(處理缺失值和異常值)、數據探索 (通過統計圖表、數據可視化工具等,初步了解數據的特徵和模式)、數據建模對數據進行深入分析,找出數據之間的關聯和趨勢。 本堂課將幫助您掌握基本的數據分析與機器學習概念,並在課堂上使用生成式AI協助進行數據處理與視覺化練習。涵蓋利用撰寫Excel VBA程式,作資料分析自動化;由ChatGPT產出Python繪圖程式碼,使用Google Colab繪製視覺化圖表等。
課程主題二:自動機器學習工具 Matlab AutoML 實作 講師: 國立暨南大學應用化學系 吳志哲教授 課程簡介: 自動機器學習(AutoML)是一種通過自動化機器學習模型的選擇、訓練和調參過程,使得非專業人員也能夠構建高效且準確的機器學習模型的技術。Matlab公司開發的AutoML工具是一款功能強大的自動機器學習軟體,旨在簡化和加速機器學習的應用流程。它提供了直觀的圖形化介面和豐富的功能,包括模型自動選擇、特徵工程、自動調參以及結果分析等。Matlab AutoML支援多種機器學習任務,如分類、回歸、效能評估等,使得用戶能夠在短時間內快速構建和部署機器學習模型,極大地降低了進行機器學習應用的門檻。 本課程特邀請國立暨南大學應用化學系的吳志哲教授,以實作方式由淺入深帶領學員熟悉Matlab AutoML的功能與操作。課程為期三小時,第一小時將介紹自動機器學習的理論基礎,幫助學員理解AutoML的核心概念和技術原理。第二小時將進行自動機器學習分類範例的實作,提供抗甲氧苯青黴素金黃色葡萄球菌(Methicillin resistant S. aureus, MRSA)與對甲氧苯青黴素敏感性之金黃色葡萄球菌(Methicillin susceptible S. aureus, MSSA) 的蛋白質質譜數據,學員將親自上手操作並體驗如何使用Matlab AutoML進行分類任務。第三小時則會展示自動機器學習在回歸任務中的應用,並預留時間進行問答交流,解答學員在實作過程中的疑問。課程內容適合對沒有程式編碼經驗和機器學習知識背景有興趣的數據科學家和相關領域的研究人員參加,期待大家踴躍報名,共同探索自動機器學習的魅力!
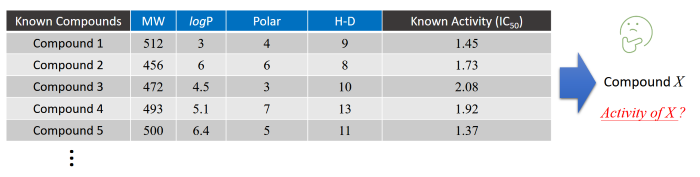
課程主題三:KNIME - No Code/Low Code資料分析平台 講師: 臺北醫學大學臨床基因體學暨蛋白質體學碩士學位學程 王三源副教授 課程簡介: 分子活性預測在藥物開發和化學研究中扮演著重要角色。通過預測分子的生物活性,可以有效地篩選出潛在的藥物候選分子,從而節省大量的實驗資源和時間。本工作坊將帶領學員從基礎開始,介紹分子活性資料庫與分子活性預測的基本原理和方法,並利用No Code/ Low Code分析工具提取分子結構資訊、進行資料前處理和模型建構等實作。
工作坊內容將以KNIME這個No Code/Low Code資料分析平台為載體,結合資料科學與化學計量學模組進行實作教學,降低學習與操作門檻,讓非程式設計背景的學員也能順利從基礎知識到實際操作,掌握分子活性預測相關技術。首先,學員將學習如何下載、安裝KNIME平台並建立工作環境,為後續的資料分析和模型建構做好準備。接著,將介紹常用的分子活性資料庫,幫助學員了解資料來源和資料結構。在分子結構描述方面,將講解如何描述分子結構,了解不同的分子描述方法及其應用,並教學如何利用KNIME計算分子的結構描述子,進行定量構效關係(QSAR)分析。接著介紹資料清理的基本方法和步驟,確保資料品質,以便進行準確的模型訓練。接下來,將介紹常用的機器學習演算法,如決策樹、隨機森林、支援向量機和神經網絡等,並解釋它們在分子活性預測中的應用。特徵篩選部分將介紹如何選擇對模型訓練最有用的分子描述子。最後,學員將學習從分子資料準備到模型建立的整個過程,並實際操作,建立並評估分子活性預測模型。期望這個主題能對相關領域之研究學者及學生在分子活性分析這個主題上有所幫助,歡迎大家踴躍參加,一起來學習利用No Code/Low Code分析工具進行分子活性預測。
2025年2月11日(星期二)AI數據分析與自動化學習工具
2025年2月12日(星期三)KNIME - No Code/Low Code資料分析平台
|
|||||||||||||||||||||||||