GMBD Bioinformatics Core 分析工具
高通量定序小RNA整合性分析平台:Deep-sequencing Small RNA Analysis Pipeline (DSAP)
DSAP 是由清華大學呂平江教授與長庚大學鄧致剛教授實驗室共同開發,針對有興趣使用高通量定序技術進行小RNA (small RNA) 研究的工作者所設計的多功能分析網站。有別於網路上其他分析小RNA的服務僅著重在少數的模式生物物種,DSAP 除可以針對一百多種已在 miRBase 登錄的物種外進行分析外,亦可選擇最接近的物作比對。
DSAP 並提供了一個友善的使用者操作界面。使用者僅需將定序產生的
RNA 序列與對應數量上傳到伺服器,並選定欲分析的生物物種或使用預設參數,即可進行全自動分析。其分析項目包含:
1. 連接子與低品質序列去除
2. 合併重覆序列
3. 非轉譯 RNA 資料庫 (Rfam) 比對
4. miRBase 資料庫比對
此外,DSAP 還能以顏色矩陣的方式,同時呈現多筆實驗資料間標準化後的 microRNA 差異表現。分析的過程與結果可透過即時互動式圖表來檢視或下載,完成分析的樣本也可透過伺服器所提供的序號加以追溯。
為了能讓使用者比較來自不同實驗平台 (Solexa、SOLiD、microarray…) 的資料,DSAP 還特地把 microRNA 差異表現的分析模組獨立出來,使用者僅需用剪貼的方式,即可將不同實驗平台的數據輸入 DSAP。最後,DSAP 還有一個最特別的功能,就是能依親緣關係顯示 microRNA 在不同物種間的分佈。
圖一:DSAP 分析工具網站
(http://dsap.cgu.edu.tw/) |
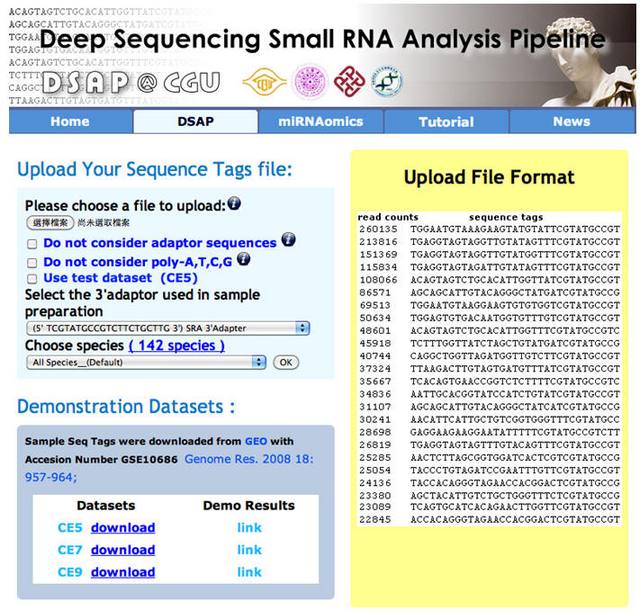
圖二:DSAP 資料上傳頁面。有關序列上傳格式與參數勾選,可點選 |
圖三:DSAP 分析流程與顯示方式 |
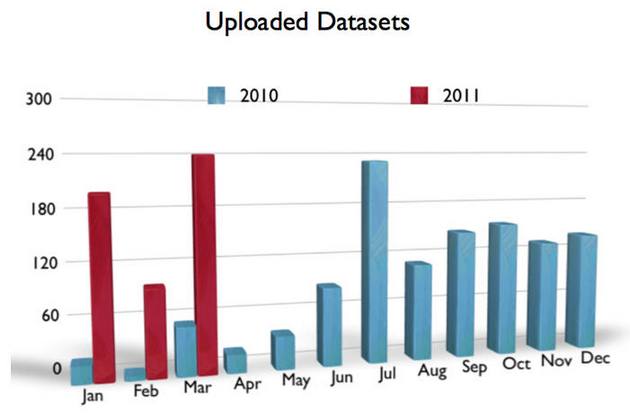
DSAP 相關論文在 2010 年 5 月刊載於知名國際期刊:核酸研究 (Nucleic Acid Research) 之後,迄今已接受超過 42 個國家,總計 1500 套以上高通量小 RNA 定序資料。除了部分來自長庚醫學院與成大醫學院,其餘多是來自於國外上傳的各種動植物之定序資料。DSAP 伺服器網址為:http://dsap.cgu.edu.tw 或 http://dsap.life.nthu.edu.tw 。為了讓全球各地的使用者能更方便快速地使用 DSAP,另有三個鏡像站將於近期上線, 分別位於香港理工大學、北京中科院基因組研究所與成大醫學院。DSAP的使用手冊,也可以從本網站中下載閱讀。
圖四:DSAP 分析伺服器使用頻率 |
圖五:DSAP 已有超過 42 個國家的使用者,進行小 RNA 資料分析 (2011年1-3月使用狀況) |