GMBD Bioinformatics Core 分析工具深度報導
單一轉錄因子下游基因大預測:The Binding Element Searching Tool (The BEST) 分析工具
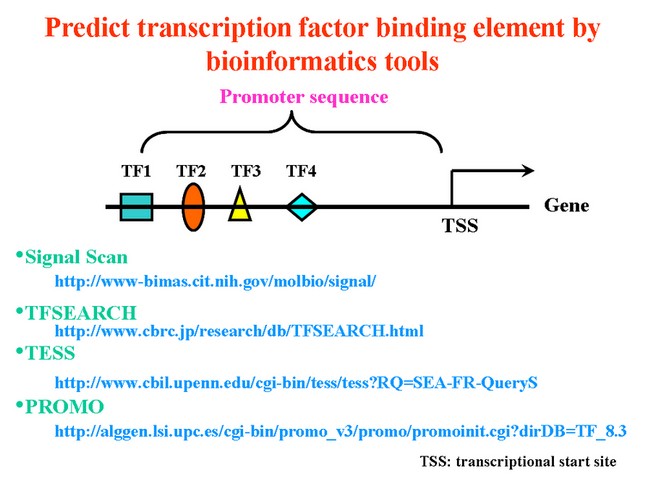
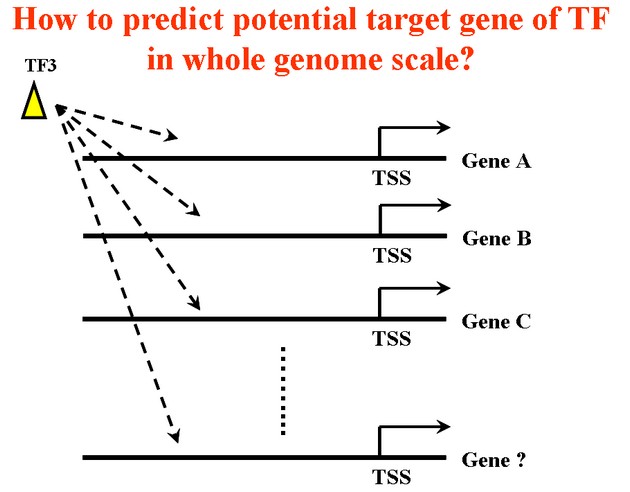
過去大家對於如何研究轉錄因子 (Transcription Factor) 對基因之表現,主要著重於多個轉錄因子可能可以調控單一個基因的表現 (圖一);也因為研究人員有這樣的需求,所以衍生出許多相關的生物資訊工具 (例如: TFSEARCH, TESS 及PROMO 等),這些工具都可以用來預測位於單一基因的啟動子上,究竟有多少可能的轉錄因子結合之位置。但是,當我們想要研究單一個的轉錄因子究竟可以調控多少基因時 (圖二),竟然發現還沒有一個已知的軟體或網站,可以幫我們達成這個目的;因此,為了達成研究目標,我們發展了「The BEST」(圖三)這個生物資訊工具。
圖一:多個轉錄因子調控單一個基因的表現 |
圖二:單一個轉錄因子調控多個基因的表現 |
圖三:The Best 分析工具網站 |
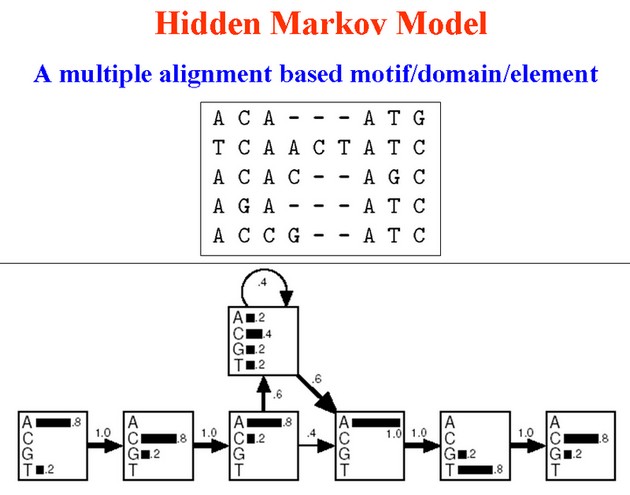
The BEST 跟一般預測轉錄因子結合位置的生物資訊工具最大的不同點,在於它是利用「隱藏式馬可夫模型 (Hidden Markov Model, HMM) 」的統計模型,來計算出轉錄因子結合位置的可能模式是如何;這跟一般以「位置加權矩陣 (Position Specific Scoring Matrix, PSSM) 」的預測工具相比,它除了考慮物件發生可能的機率之外,同時更多考慮了物件轉移的機率 (圖四);因為如此,HMM 統計模型比 PSSM 包含了更多的彈性及可能性,所以更能找出許多可能是 PSSM 所預測不出的轉錄因子結合位置。
圖四:The BEST 利用「隱藏式馬可夫模型 (Hidden Markov Model, HMM)」進行預測,具更高的準確性 |
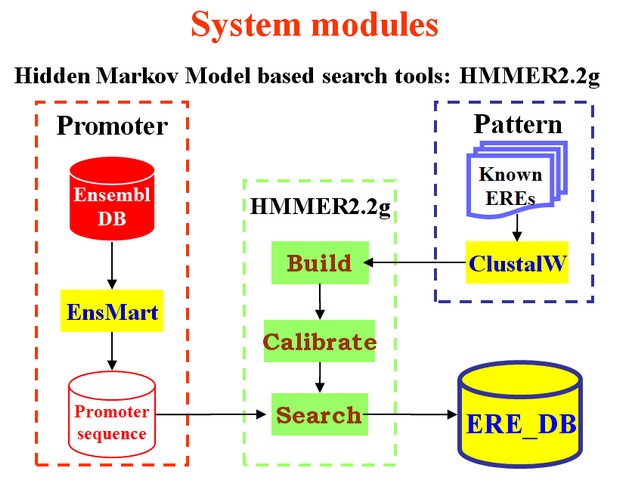
The BEST 預測系統,其開發的理論與流程如下 (圖五):首先,先利用 PUBMED 文獻搜尋系統找出 10~20 個已由實驗證實,其啟動子上確實可以讓某轉錄因子結合之基因 (例如:Estrogen receptor, ER);接著將實驗所證實之轉錄因子結合位置的序列以其核心序列 (core sequence) 為中心,擷取左右側序列共 25 base pair;接著利用 ClustalW 程式將此 10~20 個,長約 25 bp 並包含轉錄因子結合位置的序列進行多序列比對;然後將排列完的序列再以 HMM 進行運算,藉此歸納出可能的轉錄因子結合位置之可能模式 (pattern);最後用此模式 (pattern) 去搜尋人類啟動子的資料庫 (database)。由此,我們可以產生出一個可能含有轉錄因子結合位置的基因資料庫。 最終,我們可以藉由實驗方法去驗證預測出來的基因是否真的可以受此轉錄因子的調控,也找出許多之前未被發現的、可受轉錄因子調控的新穎基因。
圖五:The BEST 開發之理論與流程 |
The BEST 系統之建置,由成功大學醫學院生理所蔡少正老師所領導的研究團隊,包括分子醫學研究所孫孝芳教授、資訊管理研究所王惠嘉教授、資訊工程學系高宏宇教授以及成功大學生物資訊研究中心所組成的研發小組進行研究開發;藉由驗證此系統所預測出的結果。自 2005 年起我們已經發表數篇研究論文 (請見:publications),顯示此系統確實能預測出新的基因可受到轉錄因子的調控,也說明了此工具的高度可信度。從 2007 年至今,在全世界各地已有超過 2000 人次造訪 THE BEST 網站 (請參考使用者分布圖),充分顯示出我們所開發的工具也深受其他地區科學家之青睞。
Publications
1. Transcriptional repression of human cad gene by hypoxia inducible factor-1α. (2005) Chen KF, Lai YY, Sun HS, Tsai SJ. Nucleic Acids Res. 33(16):5190-8.
2. Induction of pyruvate dehydrogenase kinase-3 by hypoxia-inducible factor-1 promotes metabolic switch and drug resistance. (2008) Lu CW, Lin SC, Chen KF, Lai YY, Tsai SJ. J Biol Chem. 283(42):28106-14. Epub 2008.
3. Regulation of CD151 by hypoxia controls cell adhesion and metastasis in colorectal cancer. (2008) Chien CW, Lin SC, Lai YY, Lin BW, Lin SC, Lee JC, Tsai SJ. Clin Cancer Res. 14(24):8043-51. Epub 2008.