GMBD Bioinformatics Core 分析工具深度報導
細胞地圖的指南針:蛋白質在細胞位置的預測系統 (CELLO)
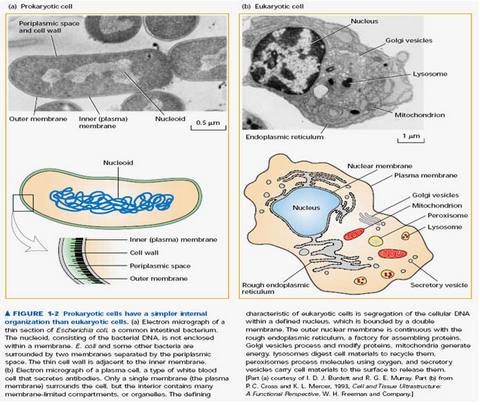
若說細胞為生命現象的基本單位,蛋白質則像是維持細胞工廠運作的工人,必須在細胞體內的不同位置 (subcellular localization) 待命,兢兢業業地分工協調著。無論是日常所見的各式動植物,或肉眼不可見之微小生物,其調控機制目前仍無法為科學界全盤了解。根據細胞結構的不同,物種可粗分為原核生物 (prokaryotes) 與真核生物 (eukaryotes) 兩大類 (見圖一),兩者最大的差別在於後者內部多了許多膜狀包覆的空間,不但將龐大的遺傳物質包裹於獨特的細胞核 (nucleus) 中,亦將許多特化的功能集中並獨立於不同的胞器進行控管,如有能量工廠之稱的粒線體 (mitochondria) 、把太陽能轉換為醣類儲存的葉綠體 (chloroplasts) 、合成蛋白質與脂質的內質網 (Endoplasmic Reticulum)、分泌蛋白質與多醣合成處理的高基氏體 (Golgi complex)、可進行水解消化功能的溶酶體 (lysosomes)、處理物質的臨時性儲存與運輸的液泡 (vacuoles)、維持細胞剛性結構以及協助細胞分裂的細胞骨架 (cytoskeleton) 等。相對於真核細胞的精細分工,原核細胞則顯得十分簡約,上述絕大多數的功能均散佈於細胞質 (cytoplasm) 或細胞膜 (plasma membrane) 上作用,雖然科學家們觀察到如此南轅北轍的構造特性,兩者卻有著運作機制上相似之處,即蛋白質分子必須到達適當的細胞位置方能開始作用。由此可知,蛋白質分子工作的位置與其負責的細胞生理功能有著密不可分的關係,而蛋白質分子本身的序列更是提供了許多重要的線索。多年前發現蛋白質的訊號胜肽 (signal peptides) 的存在正是最好的例子,可惜的是,並非所有的蛋白質都能提供科學家研究上如此有利的資訊,因此,萃取出序列中所隱含的位置訊息,以開發出計算工具將幫助科學家更快速有效的分析蛋白質功能–何時作用? 在何處作用? 對蛋白質體與基因體研究與應用將有很大的幫助。
圖一:(a) 原核細胞與(b) 真核細胞的構造差異 (Source: Lodish et.al., Molecular Cell Biology. 5th ed..New York:Freeman, 2003) |
本研究所開發的「蛋白質細胞位置預測系統」,命名為 CELLO (subCELlular LOcalization predictive system),為協助來自不同研究社群的使用者完成分析,簡化所有繁瑣的序列格式處理及操作程序,並最佳化研究過程中所需設定的各種精細參數設定,使用者僅需提供組成蛋白質之胺基酸序列或是 DNA 序列,即可在短時間內自動化、快速且準確的預測大量分子可能的細胞位置。透過計算序列上各種胺基酸特性所萃取的 n-peptide 片段組成份,建立不同的支持向量機 (Support Vector Machines) 分類器,並在許多已知的標準資料組上進行測試,均得到極佳分析結果,不但優於一般機器學習的演算法,亦省去許多組合式方法進行龐大序列資料的比對計算時間。除此之外,針對於序列和細胞位置的關係,本研究明確劃定了一個可辨識的序列相似度界限,而測試結果亦顯示不受同源性序列辨識法限制的優點。
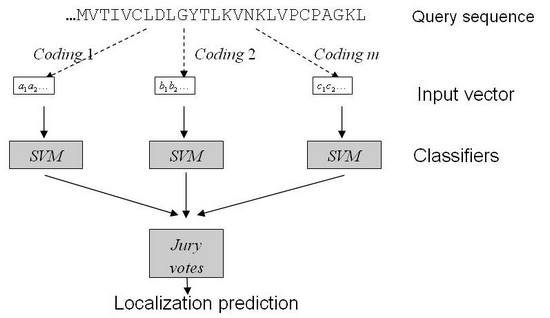
本系統根據不同細胞結構的蛋白質資料提供預測服務 (圖二),分別進行多組分類器的預測結果整合,卻非提供使用者單一位置的武斷預測,而是依照不同分類器所根據的蛋白質特性給予各細胞位置的預測分數,以相對分數區分細胞位置的可能性,提供使用者研究設計的線索。
圖二:CELLO 預測系統的流程 |
CELLO 系統之建置,乃由交通大學生物科技學院黃鎮剛院長所引領的研究團隊,包括游景盛先生、陳玉菁小姐以及陸志豪先生所組成的研發小組進行研究開發,並完成整體系統的優化與論文的撰寫。CELLO 的相關研究已發表於 2004 年的 Protein Science (13:1402-1406, 2004) 和 2006 年的 Proteins (64: 643-651, 2006) 等知名國際知名期刊,同時也被多次引用於 Developmental Cell、FEMS Microbiology reviews、Microbiology and Molecular Biology Reviews、Nature Reviews Microbiology、Nature Protocols、Nucleic Acids Research、PNAS、Bioinformatics 以及 PLoS ONE 等著名期刊的論文之中;截自 2010 年 3 月底為止,已有數萬人次使用 CELLO,以及超過 130 次的論文引用次數。說明了此工具的高度可信度和使用的便利性,對於大量 high-throughput 的蛋白質體和基因體資料分析,都具有相當的幫助。
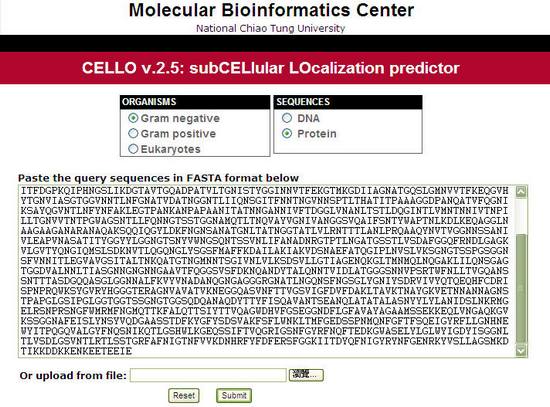
圖三:CELLO 網站介面 |
Citation:
1. Protein Science (2004), Vol. 13, p1402–1406
2. Proteins: Structure, Function and Bioinformatics, 2006, Vol. 64, p643-651
網址: http://cello.life.nctu.edu.tw
※近期使用者以 CELLO 分析,所發表的學術研究論文如下:
| 圖四:南卡大的研究團隊,在海洋微生物的鹼性磷酸化酵素 (marine bacterial alkaline phosphatases) 研究。利用 CELLO 協助分析比較大量海水樣本微生物的鹼性磷酸化酵素,進一步指出透過統計資料庫樣本蛋白質的細胞位置可知的不同微生物物種的生態指標,並可進一步由此線索探討不同微生物的相關基因調控機制。 (PNAS 2009, 106(50):21219-21223)。 |
圖五:利用 CELLO 偵測微生物 (Salmonella enterica) 蛋白。(BMC Microbiology 2010, 10:44)。 |
圖六:利用 CELLO 偵測微生物 (Helicobacter pylori) 蛋白。 (PLoS one 2008, 3:e3510)。 |
圖七:利用 CELLO 偵測微生物 (Vibrio sp.) 蛋白。 (Applied and Environmental Microbiology 2007, 73:2825-2831)。 |
圖八:利用 CELLO 偵測真核生物的細胞核蛋白。 (Genome Biology 2008, 9:R15)。 |