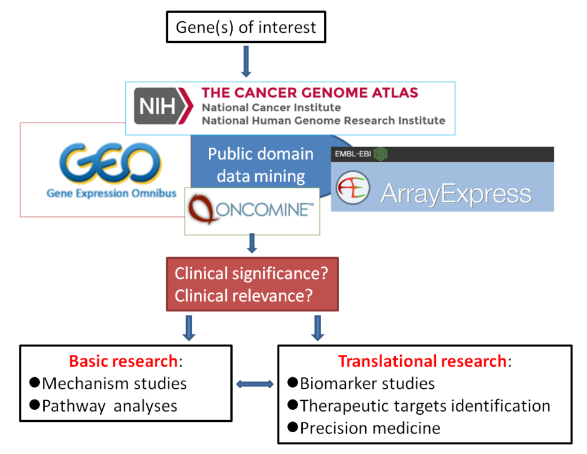
利用癌症公用基因表現資料庫探索癌症基因的臨床意義
如果讀者正著手研究一個感興趣的基因,正在猜想此基因會在某種癌細胞扮演重要的功能、角色,那麼對多數人而言,傳統的作法是先利用癌症細胞株進行in vitro實驗,例如siRNA默化或強迫該基因表現,觀察癌細胞的行為、特徵是否改變,而臨床意義,就只能利用數量有限的珍貴臨床檢體進行qPCR基因表現定量分析,或者是以IHC(免疫組織染色)進行蛋白質表現分析。
先別急著投入這些昂貴的實驗吧!轉譯醫學暨生技研發之生物資訊核心設施(簡稱TMBD核心),將提供您更好的選擇!我們可以換個方式問:你(妳)感興趣的基因是否已被包含(埋沒)在某些基因組剖析(genomic profiling)的實驗裡而待開採?若將相關的資料挖掘出來,是否可以顯示出該基因的癌生物學及臨床意義?例如:相較於正常組織是否過度表現或表現低下?基因表現與癌症存活、預後是否有關? 基因表現與藥物反應或敏感度是否相關?哪些已知的癌症基因和該基因有相似或相反的表現圖譜?基因表現特徵在各類癌症是否一致? 基因表現特徵是否有癌症種類之專一性?
難以否認的,臨床意義(clinical significance)與臨床相關性(clinical relevance)是轉譯醫學研究(translational medicine research) 的基本元素。這些元素是讓基礎生物醫學研究通往臨床應用的重要橋樑,而這些元素當然是以對臨床檢體、樣本等的觀察與分析為基礎所建立的。然而寶貴的臨床檢體庫常不可多得,誰又願意提供這些資源做沒有把握的少數基因剖析與分析?所幸,在後基因組時代(post-genome era),由前人所公開分享出來,而累積在公用資料庫(例如GEO及ArrayExpress)或是大的研究計劃(如TCGA)的這些被埋沒的基因表現數據,TMBD核心因此得以資料採礦(data mining)的方式,建構上述基礎研究與轉譯研究的橋樑。
如果讀者已有一些in vitro細胞株的實驗數據,可以嘗試利用以癌症臨床樣本或細胞株所建立的癌症基因表現資料庫進行資料採礦,除了可以檢驗預設假說的一致性(consistency)與完整性(integrity),更可以補強研究的臨床意義與臨床相關性。TMBD核心及國衛院癌研所張憶壽博士、江士昇博士與合作團隊的論文發表,已有不少的例子可供讀者參考。例如:本團隊在SOX9基因在肺腺癌臨床檢體的表現分析(Clinical Cancer Research, 2010)以及SOX2在肺鱗狀細胞癌調控BMP4基因表現的臨床與生物意義(International Journal of Cancer, 2014), 國衛院癌研所李岳倫副研究員對GDF10協同TGFBR3調控口腔癌細胞EMT的訊息傳遞路徑分析與臨床意義研究(Molecular Carcinogenesis, 2015),以及國衛院細胞與系統醫學研究所褚志斌副研究員對AKT3在攝護腺癌細胞中調控AKT、BRAF、TSC1/TSC2等的癌症檢體基因表現資料庫資料採礦分析 (Oncotarget, 2015)等。倘若讀者仍在研究規劃(study design)階段,如果可以利用這些資料庫,先探索你感興趣基因的臨床特徵與意義,或許可以使讀者更具信心深入生物實驗,當然也可能讓你打消沒有把握的念頭,節省一筆研究經費、資源和寶貴的時間,去找尋更有意義、價值的癌症相關基因。
除了上述對癌生物學研究者的強大支援外,我們也在建立癌症公用基因表現資料庫探索平台的過程中,也發現一些此類方法的限制與缺點。TMBD核心未來將舉辦一系列的課程,分享如何有效利用癌症公用基因表現資料庫的經驗,敬請有興趣的癌症研究同好密切注意。