● MicroRNA資料分析客製化服務
微小核醣核酸MicroRNA (miRNA),是一個可由生物體內自行合成的小片段核醣核酸,長度約為22鹼基對(base pairs)。miRNA經由DNA轉錄而來,但是無法轉譯成為蛋白質,屬於非編碼核醣核酸 (non-coding RNA);可以透過與標靶基因轉錄出的訊息核醣核醣核酸(mRNA)結合,使其標靶基因之訊息核醣核酸被降解,或者可以透過阻擋蛋白質轉譯,進而抑制基因的表現。第一個miRNA於1993年時在線蟲中被發現後,大約在2003年miRNA開始被大量的研究,直到現在相關研究呈指數般的成長速度在增加,目前在人類中已發現超過1800個miRNA的前驅物(precursor miRNA),以及2500個以上的成熟miRNA(mature miRNA)。交通大學黃憲達教授所帶領的研究團隊也投入相關的研究(如圖一),例如:miRStart、miRTarBase、miRTar …等。
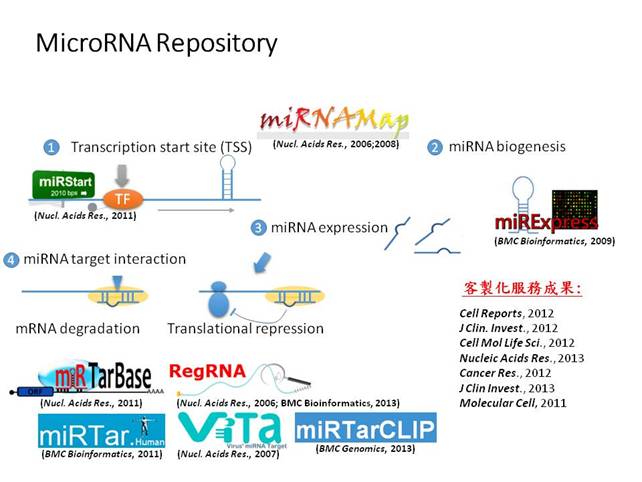
圖一: MicroRNA 相關研究及客製化服務成果,從miRNA基因的轉錄起始點、miRNA表現量到各式各樣的miRNA標靶基因分析工具及資料庫,另外目前也已經有許多客製化服務的成果已發表在很好的期刊。 |
目前所研發的成果包括,從miRNA的轉錄起始位置的找尋及預測開始(miRStart),利用實驗資料計算分析miRNA的表現量(如 miRExpress, miRNAMap),以了解miRNA會在特殊環境下被啟動,而通常這些高表現的miRNA很可能是調控基因表現量的關鍵。另外蒐集已知或預測的miRNA target interaction (MTI) 並建立相關資料庫或分析工具 (如 miRTarBase, miRTar, miRTarCLIP, miRNAMap),對於了解整體miRNA調控網路的研究是極具有幫助的。由圖一可以清楚的了解我們目前對於miRNA在每個階段的相關研究,從最上游miRNA 如何被啟動,直至下游miRNA影響基因表現量之分析、註解工具和資料庫,其中包含許多分析技術及經驗,可以提供給生物學家從不同的角度來進一步了解以及分析miRNA,例如,分析癌症、心血管疾病、免疫及病毒與miRNA之間的關係。
● MicroRNA 分析工具和資料庫
miRStart: 微小核醣核酸轉錄起始點資料庫。
miRTarBase: 蒐集經實驗驗證的miRNA-target交互作用資料。
miRTar: 多角度分析miRNA和調控基因關係,及其路徑的工具。
miRExpress: 藉由高通量定序資料進行微核醣核酸表現分析體。
miRNAMap: 微小核醣核酸在後生動物基因組中的地圖
RNALogo: 呈現RNA二級結構與保留序列之繪圖工具。
ViTa: 病毒miRNA相關文獻以及目標基因資料庫。
miRTarCLIP: 微小核醣核酸目標位置分析工具。
RegRNA 2.0: 利用整合性計算平台找尋Regulatory RNA motifs及研究其功能。
● miRTarBase 4.0:收集經實驗驗證的miRNA-target交互作用資料庫
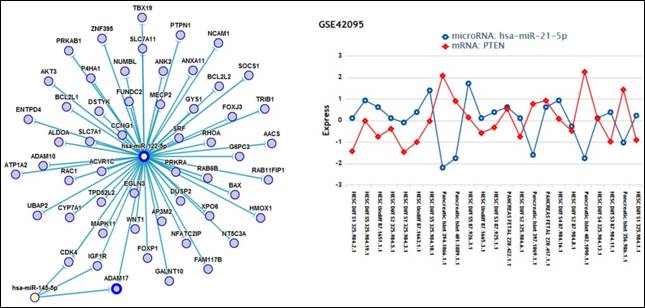
miRTarBase是一個專門收集經實驗驗證的miRNA與標靶基因交互作用的資料庫,透過資料庫可查詢到miRNA會調控哪些標靶基因,也可以得知感興趣的基因會被那些miRNA所調控,而這些資料都是經過不同的實驗驗證過。另外miRTarBase也提供詳細的miRNA與標靶基因的資料,例如,miRNA於標靶基因上,明確的標靶位置,若文獻沒提供,我們也會利用標靶基因預測軟體找出可能的標靶位置。當然也會提供文獻出處和其描述此標靶基因關鍵的句子。此資料庫的內容是經由人工閱讀文獻後再匯整而成,大約兩到三個月就會更新一次,以提供最新最完整的資料。miRTarBase release 1.0於2011年發表於Nucleic Acids Res (2011),這幾年間除了繼續更新miRNA-target相關資訊,我們最近更做出一些重大更新(表一)並提供miRNA-target 網路關係圖及miRNA-target 基因表現的功能(圖二)。miRTarBase網址為
http://miRTarBase.mbc.nctu.edu.tw/
|
Features |
miRTarBase 1.0 |
miRTarBase 4.0 |
|
Known
miRNA entry |
miRBase
(version 14) |
miRBase
(version 20) |
|
Species |
17
species |
17
species |
|
Curated
miRNA-target interactions |
3,576
MTIs |
34,083
MTIs |
|
MTIs were
validated by experimental
technology |
Reporter
assay, western blot,
Northern blot, qRT-PCR,
microarray, pSILAC |
Reporter
assay, western blot,
Northern blot, qRT-PCR,
microarray, pSILAC, NGS
(CLIP-Seq and Degradome-Seq) |
|
Number of
MTIs validated by “Reporter
assay” or “Western blot” |
2,207
MTIs |
4,558
MTIs |
|
miRNA
expression profile |
None |
NCBI GEO
(21 datasets) |
|
Target
gene expression profile |
None |
NCBI GEO
(21 datasets) |
|
Relationship between miRNA
and target genes |
None |
Analysis
of the expression profiles
between miRNA and its target
genes |
|
miRNA
regulatory network |
None |
First-order neighbors of
miRNA and target gene. |
|
Disease
information |
None |
187 diseases |
|
Upgrade reminder
service |
None |
Yes |
|
Error report system |
None |
Yes |
|
Graphical
visualization |
miRNA, secondary structure, known and novel
miRNA target sites, functional and
non-functional MTIs, experimental conditions |
miRNA, secondary structure, known and novel
miRNA target sites, functional and
non-functional MTIs, experimental
conditions, miRNA-target network, miRNA-target
expression profile, miRNA disease, upgrade
reminder service, error report system, user
feedback service |
表一:miRTarBase 4.0主要更新功能。
圖二: miRNA-target網路關係圖及基因表現。 |
Publication:
S.D. Hsu, F.M. Lin, W.Y. Wu, C. Liang, C.J. Lee, C.Y. Huang, A.P. Tsou and H.D. Huang* (2011) "miRTarBase: a database curates experimentally validated microRNA-target interactions" Nucleic Acids Research, Vol.39, pp. D163-9.
● miRTarCLIP: 藉由高通量免疫沉澱法辨識微小核醣核酸標靶基因位置
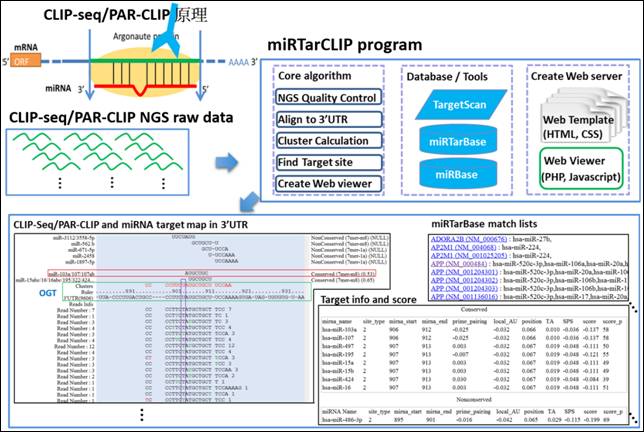
微小核醣核醣核酸(microRNA)調控了許多重要的基因表現,傳統利用報導基因方法一個一個來驗證miRNA-標靶基因的方法非常耗時與耗人力。當今已有高通量定序的方法(CLIP-seq and PAR-CLIP)可把所有的miRNA-標靶基因的位置全部辨識出,而miRTarCLIP就是基於此高通量技術所發展出來的重要分析工具。未來生物學家想要研究某個miRNA所控管的所有基因的話,只要利用此高通量定序實驗配合miRTarCLIP工具的分析,就可以把所有的miRNA標靶基因的關係全部找出。http://miRTarCLIP.mbc.nctu.edu.tw
圖三: CLIP-seq/PAR-CLIP 原理及miRTarCLIP 分析流程 |
Publication:
C.H. Chou, F.M. Lin, M.T. Chou, S.D. Hsu, T.H. Chang, S.L. Weng, S. Shrestha, C.C. Hsiao, J.H. Hung* and H.D. Huang* (2013) A computational approach for identifying microRNA-target interactions using high-throughput CLIP and PAR-CLIP sequencing" BMC Genomics, Vol. 14, Suppl. 1: S2.
● RegRNA 2.0: 利用整合性計算平台找尋Regulatory RNA motifs及研究其功能
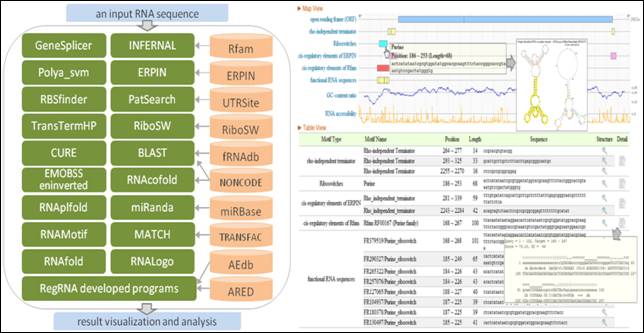
RegRNA 為全世界最完整收錄 Regulatory RNA motifs 的系統。其中牽涉了 Gene transcriptional regulation、post-transcriptional regulation、RNA stability 和 miRNA regulation 等機制,對於 Regulatory RNA 的控制角色和功能的研究,有著極大的助益。RegRNA資料庫的建置為完成『RNA藥物標的找尋系統 (RNA Drug Targets Hunting System)』的第一個重要的階段性目標。基因體研究提供了許多機會,讓科學家能夠以蛋白質和RNA為目標,進行藥物設計研究。某些RNA區段提供了一個新的藥物設計標的。http://RegRNA2.mbc.nctu.edu.tw/
圖四:系統分析流程圖及圖形化介面 |
Publication:
1. T.H. Chang, H.Y. Huang, J. B.K. Hsu, S.L. Weng, J.T. Horng* and H.D. Huang* (2013) "An enhanced computational platform for investigating the roles of regulatory RNA and for identifying functional RNA motifs" BMC Bioinformatics, Vol. 14, Suppl. 2: S4
2. H.Y. Huang, C.H. Chien, and K.H. Jen, H.D. Huang* (2006) "RegRNA: A regulatory RNA motifs and elements finder" Nucleic Acids Research, Vol. 34, W429-W434
● 客製化服務成果
1. MicroRNA-122在肝癌中扮演重要的角色
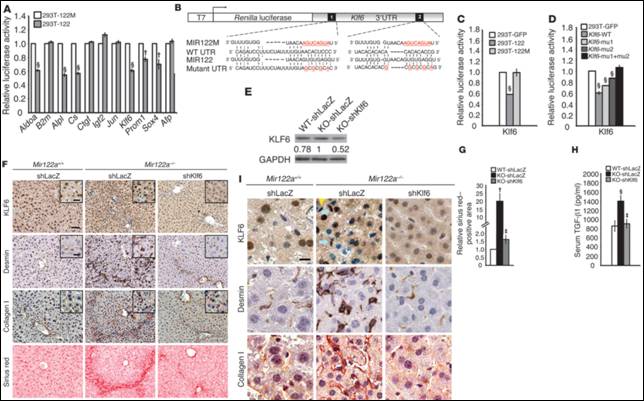
在高移動性的肝癌細胞株中大量表現 miR-122,能夠有效的減少腫瘤生成、血管新生以及防止腫瘤之肝內轉移,因此 miR-122 為肝臟組織中一個抑癌微核醣核酸(tumor suppressor miRNA)。利用 mir-122 基因剔除老鼠(mir-122 -/-)。初步的結果顯示,mir-122 -/-鼠可以正常的生活及生育,但因脂肪代謝的異常,形成類似人類的「非酒精性脂肪肝」,依序的產生肝炎、肝纖維化以及肝癌等病變,類似人類肝臟疾病的病程。我們根據服務對象的需求設計了一系列的客製化分析平台,利用微陣列表現晶片、配合生物資訊預測及實驗驗證了數個目標基因。其中一個目標基因 Klf6為主控纖維化的轉錄因子,並且會引起肝纖維化 (圖五)。
圖五:Klf6為miR-122的目標基因並會引起肝纖維化。 |
Publication:
W.C. Tsai, S.D. Hsu, C.S. Hsu, T.C. Lai, S.J. Chen, R. Shen, Yi Huang, H.C. Chen, C.H. Lee, T.F. Tsai, M.T. Hsu, J.C. Wu, H.D. Huang*, M.S. Shiao*, M. Hsiao*, A.P. Tsou* (2012) "MicroRNA-122 plays a critical role in liver homeostasis and hepatocarcinogenesis" Journal of Clinical Investigation , Vol. 122, No. 8, pp. 2884-97.
2. 微小核醣核酸與C型肝炎病毒基因體的交互作用與功能性探討
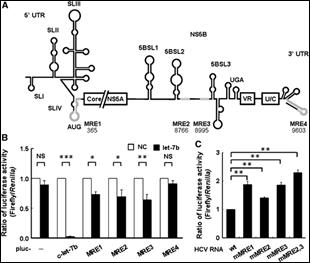
C 型肝炎病毒感染可造成肝硬化甚至肝癌,為全球性的重要議題。以干擾素結合 Ribavirin 為目前的標準治療策略,然而約有一半的患者並不能被完全根治。因此,若能抑制調控C 型肝炎病毒感染的宿主細胞因子,將可發展成為新的預防或治療策略。除了宿主蛋白外,微小核醣核酸已被發現可以調控 C 型肝炎病毒感染,也因此提供了另一個可行的抗 C 型肝炎感染的宿主標的。我們以生物資訊分析肝臟表達較高的微小核醣核酸發現除了已知可以調控C 型肝炎病毒感染的 miR-122 外,微小核醣核酸 let-7b 與 miR-16 有機會可與 C 型肝炎病毒基因體結合。本項研究進一步以 C 型肝炎病毒複製細胞進行實驗,發現微小核醣核酸let-7b 可以抑制 C 型肝炎病毒 RNA 含量 (圖六),證實我們在生物資訊上的預測結果,並暗示微小核醣核酸 let-7b 可能是 C 型肝炎病毒感染的負調控因子。
圖六:let-7b 標的基因位點座落於 HCV基因體中的NS5B序列上。 |
Publication:
J.C. Cheng, Y.J. Yeh, C.P. Tseng, S.D. Hsu, Y.L. Chang, N. Sakamoto, H.D. Huang* (2012) "Let-7b is a novel regulator of hepatitis C virus replication" Cellular and Molecular Life Sciences, Vol. 69, No. 15, pp. 2621-33.
3. miR-103/107藉由結合轉移抑制基因DAPK 和KLF4 促進大腸直腸癌的轉移
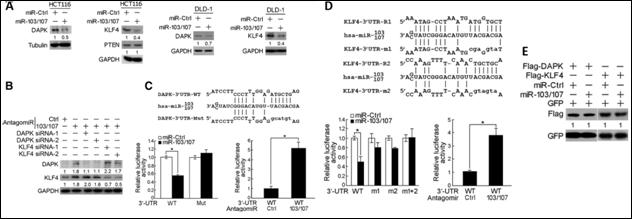
目前有越來越多的證據支持 miRNA 對於癌症惡化影響。我們以生物資訊及統計分析在大腸直腸癌(colorectal cancer, CRC)的研究中 miR-103/107 藉由抑制基因DAPK 和KLF4促進(造成)腫瘤轉移。我們發現 miR-103/107 高表現和大腸直腸癌症轉移、以及較差的預後相關。在 CRC 中 miR-103/107 和癌症轉移基因 DAPK 和 KLF4 結合後,造成細胞死亡率和細胞與基質附著上升,同時會降低細胞與細胞間附著以及上皮細胞指標表現量。在缺氧的情況下 miR-103/107 表達增加,因而造成 DAPK 和 KLF4 表現量的下降,誘導細胞轉移和侵襲。在小鼠大腸直腸癌的模型中,miR-103/107 的過度表達,造成肝臟的局部浸潤和肝轉移,而重新表達 DAPK 或 KLF4 則可以抑制這樣的現象。miR-103/107 同樣也可以藉由 DAPK 和 KLF4 使的轉移位置的細胞發生同樣的現象。在臨床上, miR-103/107 高表現量和 DAPK 以及 KLF4 低的表現量以及 CRC 患者淋巴結和遠處轉移的程度,可以視為轉移復發和不佳的預後一個指標。因此,研究結果指出,在 CRC 中經由 miR-103/107 抑制 DAPK 和 KLF4 的表現會促進癌症的轉移,可能是部分缺氧導致刺激腫瘤轉移。破壞這種調節機制的策略,可以阻止CRC的轉移。在本研究中,利用生物資訊及統計分析微陣列資料並結合 miRTarBase 資料庫以及預測工具提供miRNA標的基因的候選清單,並藉由系列的實驗驗證此一機制存在於活體之中。
圖七:miR-103/107和預測標的基因 DAPK 以及 KLF4結合位置的相關驗證實驗 |
Publication:
H.Y. Chen, Y.M. Lin, H.C. Chung, Y.D. Lang, C.J. Lin, J. Huang, W.C. Wang, F.M. Lin, Z. Chen, H.D. Huang, J. Y.J. Shyy, J.T. Liang, and R.H. Chen* (2012) "miR-103/107 promote metastasis of colorectal cancer by targeting the metastasis suppressors DAPK and KLF4" Cancer Research, Vol. 72, No. 14, pp. 3631-41.
4. 缺氧性高表現之微核醣核酸會抑制Argonaute1進而促進血管增生
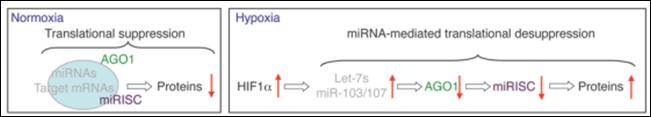
藉由miRExpress分析高通量定序的 miRNA資料發現,細胞在缺氧狀況下會大量表現缺氧反應高表現之miRNA (HRMs) ,進一步利用miRNA-標靶基因工具及資料庫如miRTar, miRTarBase分析,其miRNA包含Let-7及 miR-103/107,缺氧狀況下基因HIF1a將高度表現,HIF1a高度表現下將刺激HRMs高度表現,該miRNA被預測會抑制argonaute 1 (AGO1) 表現,以我們分析 miRNA得到的結果進行實驗觀察,發現 AGO1被抑制會造成VEGF基因表現量增加,此外抑制HRMs表現或提升AGO1表現量讓組織產生缺氧性血管增生,再者於AGO1剔除之細胞組織下於正常供氧狀況下會有血管增生現象,在異種移植實驗及人類癌組織也有發現類似的現象,此AGO1調控VEGF之機制可能是造成癌症血管增及較差的癌症預後之主因,本研究的發現將可為癌症治療提供新的抗血管新之基因標靶目標。缺氧所造成的的控模型圖示於圖八。
圖八:左圖:正常供氧狀態AGO1正常表現抑制血管增生相關之蛋白質表現。右圖:缺氧狀態下HIF1a高度表現調控Let-7s及miR-103/107高度表現,該miRNA抑制AGO1生成間接造成血管增生調控基因高度表現。 |
Publication:
Z. Chen, W.C. Wang, H. Xiao, Y.T. Wang, F.M. Lin, W. Sun, T.C. Lai, Y.H. Jan, Y.S. Li, X. Cui, T. Fang, H. Zhao, C. Padmanabhan, R. Sun, D.L. Wang, H. Jin, H.D. Huang, M. Hsiao and J. Y.J. Shyy1* (2013) “Angiogenesis Enhancement via MicroRNA Targeting of Argonaute 1: A Hypoxia-induced Translational De-suppression Mechanism” Journal of Clinical Investigation, Vol. 123, No. 3, pp. 1057-67.