TBI - microRNA分析工具
微小核醣核酸MicroRNA (miRNA),是一個可由生物體內自行合成的小片段核醣核酸,長度約為22鹼基對(base pair),miRNA屬於非編碼核醣核酸 (不會編碼出任何蛋白質);可以透過與標靶基因轉錄出的訊息核醣核醣核酸(mRNA)結合,使其標靶基因之訊息核醣核醣核酸被降解,或者可以透過阻擋蛋白質轉譯,進而抑制基因的表現。從第一個miRNA於1993年時在線蟲中被發現後,大約在2003年miRNA開始被大量的研究,直到現在相關研究呈指數般的成長速度在增加。我們(由交通大學黃憲達教授所帶領的研究團隊)也投入相關的研究(如圖表一),例如:miRSTart、miRTarBase、miRTar …等。
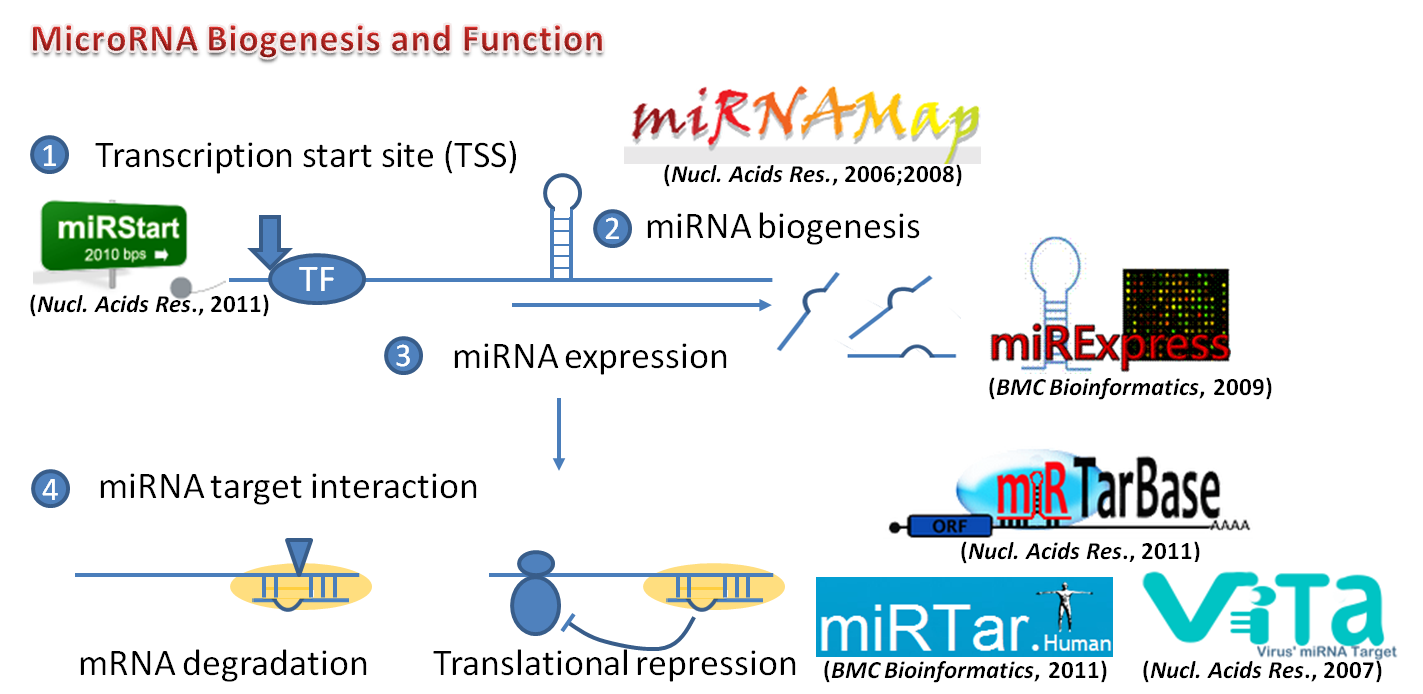
圖表一: MicroRNA 相關研究,從miRNA基因的轉錄起始點、miRNA表現量到miRNA標靶基因分析。 |
我們從miRNA的轉錄起始位置的找尋及預測開始(如 miRStart),再來從實驗資料計算分析miRNA的表現量(如 miRExpress, miRNAMap),以了解miRNA會在特殊環境下被啟動,通常這些高表現的miRNA很可能是調控基因表現量的關鍵。另外蒐集已知或預測的miRNA target interaction (MTI)並建立相關資料庫或分析工具(如 miRTarBase, miRTar, miRNAMap),對於miRNA的調控網路研究是有幫助的。由圖表一可以清楚的了解我們對於miRNA每個階段的相關研究,從最上游miRNA 如何被啟動,至下游的miRNA影響基因表現量之分析工具和資料庫,其中包含許多分析技術及經驗,可以提供給生物學家從不同的角度來進一步分析miRNA,例如,分析癌症、心血管疾病、免疫及病毒與miRNA之間的關係。
● miRStart (Nucl. Acids Res., 2011)
miRStart –微小核醣核酸轉錄起始點資料庫
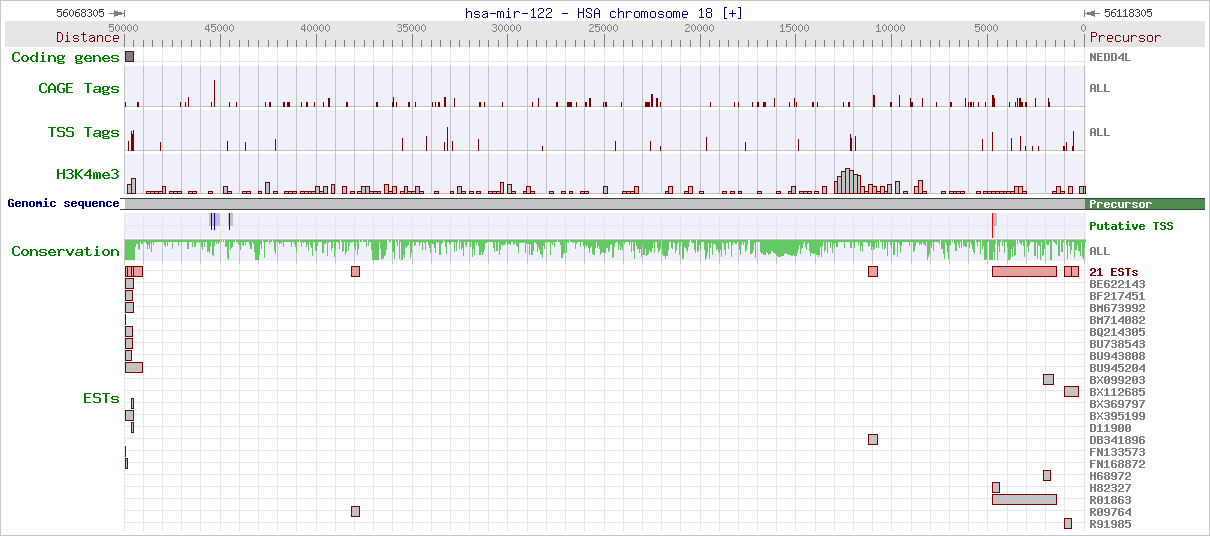
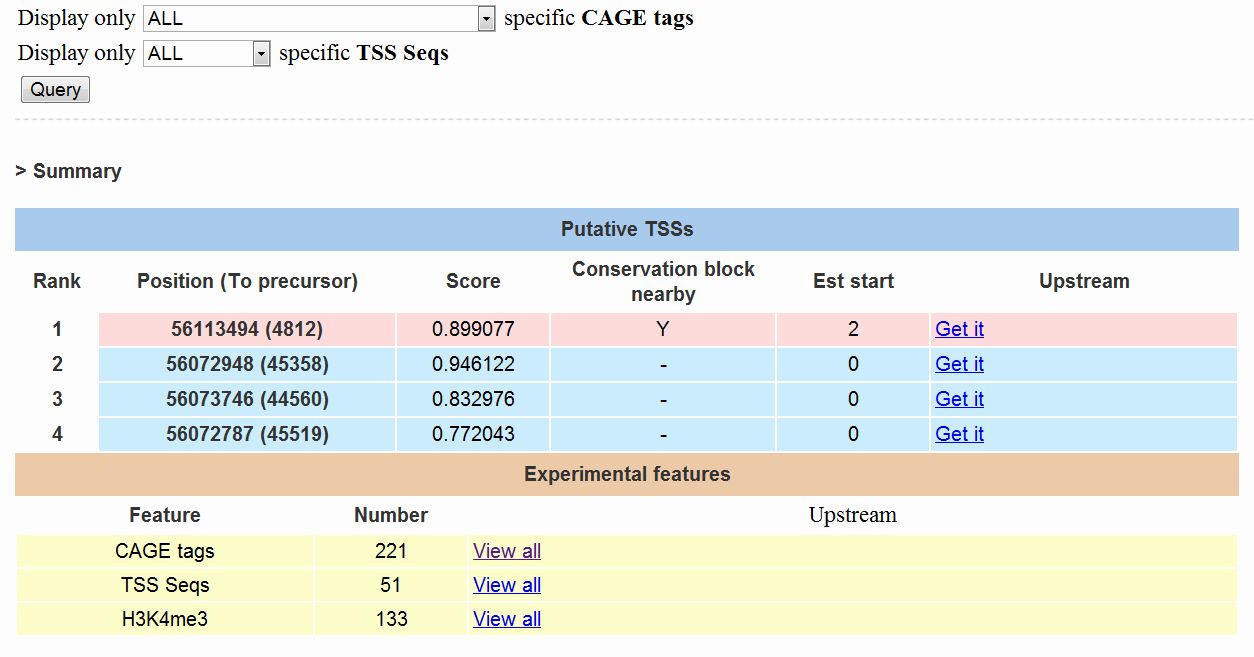
微小核醣核醣核酸(microRNA)在基因表現上扮演了重要的角色,藉由與標靶基因之訊息核醣核醣核酸(mRNA)之3端非編碼區(3’UTR)的結合,來中止標靶基因轉譯成蛋白質,進而影響後轉錄期各種不同的生物機制,據估計在人類基因中有相當大的比例受到microRNA調控。因此,了解microRNA被轉錄的條件與時機對於進一步了解有microRNA介入的基因調控網路是非常重要的。有鑑於大部份現有用於預測microRNA轉錄起始點(Translation start site)的工具缺乏足夠的實驗証據,我們整合了CAGE (cap analysis of gene expression) tags、 TSS Seq以及H3K4me3基因起始點染色質特徵(chromatin signature)等直接的生物實驗証據作為預測人類microRNA轉錄起始點的基礎資源。此資料庫建立了847個人類microRNA的轉錄起始位點資訊,以圖型化的方式表現各microRNA precursor序列上游的各種實驗線索如圖表二,並提供各個預測轉錄起始點的上游序列如圖表三,以便研究人員選擇以及進一步的運用。miRStart的網址為 http://mirstart.mbc.nctu.edu.tw/
Publication:
1. Chia-Hung Chien, Yi-Ming Sun, Wen-Chi Chang, Pei-Yun Chiang-Hsieh, Tzong-Yi Lee, Wei-Chih Tsai, Jorng-Tzong Horng, Ann-Ping Tsou, and Hsien-Da Huang (2011) Identifying transcriptional start sites of human microRNAs based on high-throughput sequencing data. Nucleic Acids Research, Vol. 39, No. 21, 9345-9356
圖表二: 圖形化表現miRNA precursor序列上游的各實驗結果之註解。 |
圖表三: 以表格方式呈現各實驗結果之TSS位置和序列的下載。 |
● miTarBase (Nucl. Acids Res., 2011)
miRTarBase: 收集經實驗驗證的miRNA-target交互作用資料庫
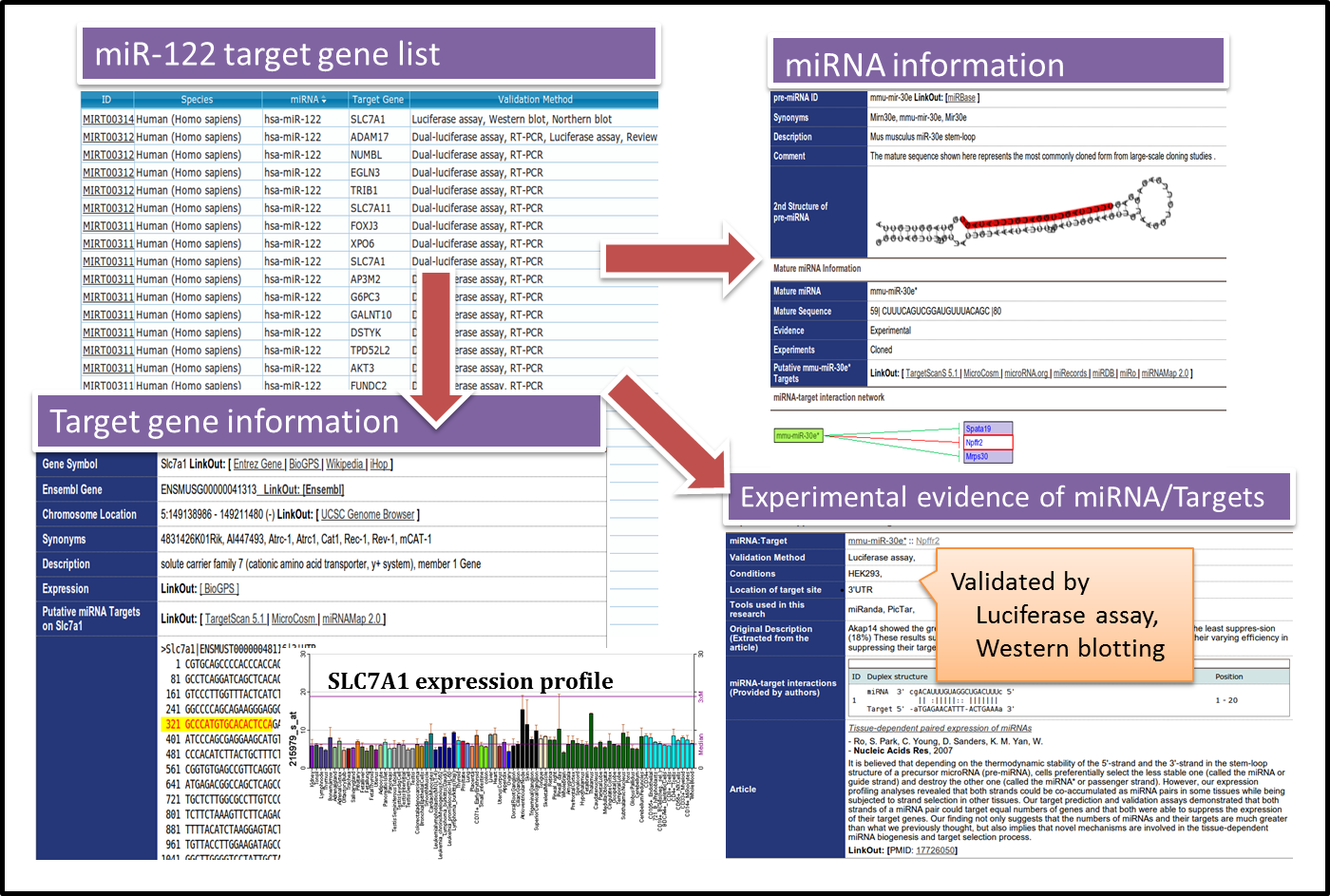
miRTarBase是一個專門收集經實驗驗證的miRNA與標靶基因交互作用的資料庫,透過資料庫可查詢到miRNA會調控哪些標靶基因,也可以得知感興趣的基因會被那些miRNA所調控,而這些資料都是經過不同的實驗驗證過 (如圖表四所示),另外miRTarBase也提供詳細的miRNA與標靶基因的資料,例如,miRNA於標靶基因上,明確的標靶位置,若文獻沒提供,我們也會利用標靶基因預測軟體找出可能的標靶位置。當然也會提供文獻出處和其描述此標靶基因關鍵的句子。此資料庫的內容是經由人工閱讀文獻後再匯整而成,大約兩到三個月就會更新一次,以提供最新最完整的資料。miRTarBase網址為 http://mirtarbase.mbc.nctu.edu.tw/
Publication:
1. Hsu SD, Lin FM, Wu WY, Liang C, Huang WC, Chan WL, Tsai WT, Chen GZ, Lee CJ, Chiu CM et al: miRTarBase: a database curates experimentally validated microRNA-target interactions. Nucleic Acids Res 2011, 39(Database issue):D163-169.
圖表四: miRTarBase的圖型化呈現介面。 |
● miRTar (BMC Bioinformatics, 2011)
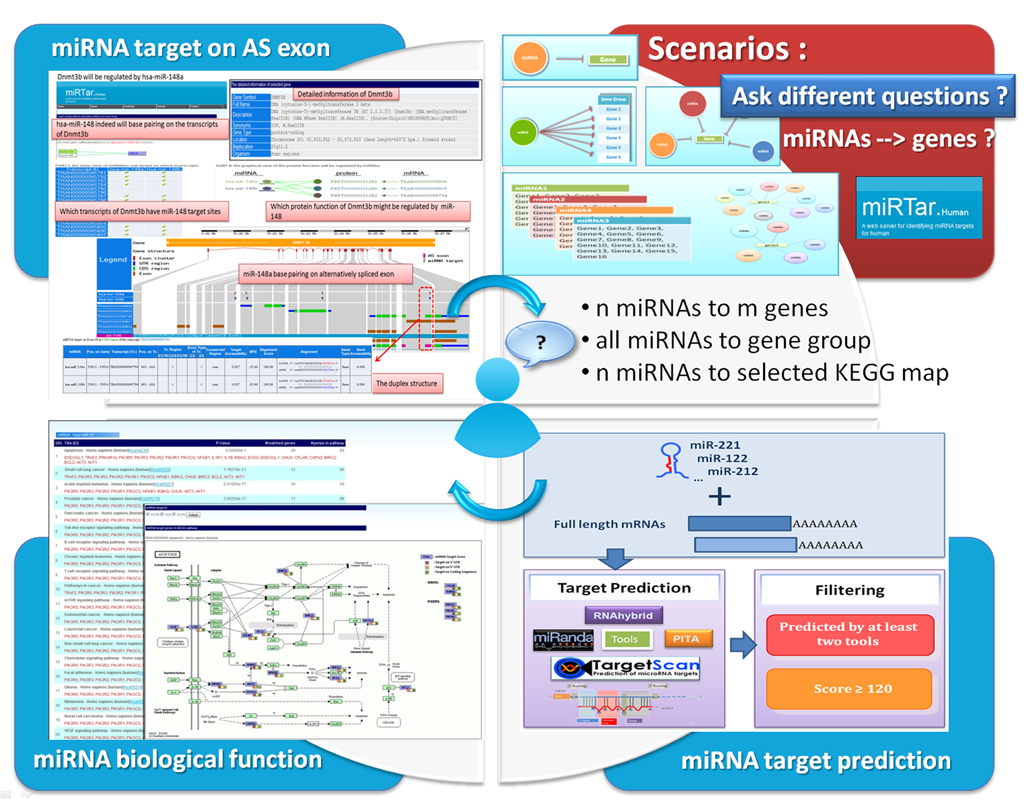
miRTar, 是一個整合性的分析系統,主要可以提供多種分析角度讓使用者可以依據不同的需求而得到微小核醣核酸(miRNA)和標靶基因表現的關係(例如:生物學家所感興趣的基因,可能會被哪些微小核醣核酸所調控...等),並進而說明微小核醣核酸於生物代謝路徑(biological pathway)中扮演可能的角色。此分析系統有三個主要特色:
I. 多個分析角度如以下所列 (如圖表五右上角所示)
● miRNA : 1 gene
● miRNA : N genes
● N miRNAs : 1 gene
● N miRNAs : M genes
● All miRNAs : N genes
● N miRNAs : genes involved in a pathway
II. 透過一群基因共同參與特定的生物代謝路徑(KEGG Pathway),且可同時被某一微小核醣核所調控,進而推測此微小核醣核酸可能的生物功能。(如圖表五左下角所示)
III. 圖形化標示出微小核醣核酸於標靶基因上的位置(5端非編碼區域、編碼區域、3端非編碼區堿),且註解標靶位置恰好座落在選擇性剪接的外顯子(因為此標靶位置可能因為選擇性剪接外顯子的移除,而使得基因表現不會受此微小核醣核酸的影響而被抑制)。(如圖表五左上角所示)
在微小核醣核酸標靶預測的研究領域中,我們開發此整合性資源miRTar,可以讓生物學家透過簡單的介面操作,方便探討已知的或可能的微小核醣核酸與標靶基因之間的調控關係以及微小核醣核酸所扮演的生物功能。miRTar網址為http://miRTar.mbc.nctu.edu.tw
Publication:
1. Hsu JB, Chiu CM, Hsu SD, Huang WY, hien CH, Lee TY, Huang HD: miRTar: an integrated system for identifying miRNA-target interactions in human. BMC Bioinformatics 2011, 12:300.
圖表五: miRTar分析系統之概念圖。 |
● miRExpress (BMC Bioinformatics, 2009)
miRExpress: 藉由分析高通量定序資料進行微核醣核酸表現定量
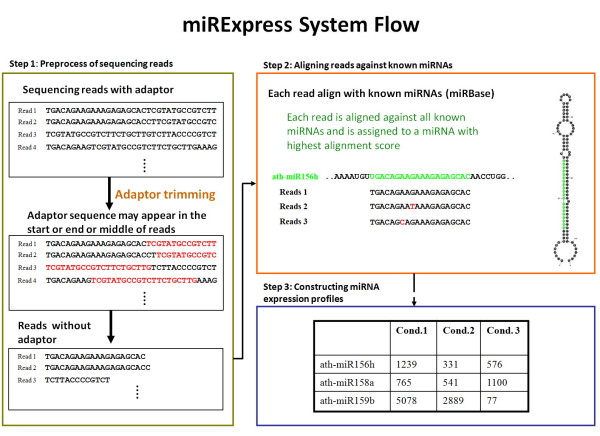
miRNA為一長度19~25核醣核酸序列,主要在於調控基因的表現,miRNA與3’UTR結合,進而抑制轉錄後基因表現,在調控基因表現、細胞周期調控、生物體發展等各方扮演重要角色。近年來次是代定序技術蓬勃發展,單位定序量的價格不斷下降,日益普及,借由高通量定序小核醣核酸,可快速取得細胞體內微小核酸的資訊,小核酸資訊經由miRExpress與miRBase微核醣核酸資料庫進行分析比對後可快速取得各miRNA表現量,其序列分析方式如圖表六所示,先進行Adaptor序列除去,然後對各miRNA相同的序列進行數量累積以便或取各miRNA表現量。miRExpress現為一免費分析軟體,可供各方學者下載使用,下載網址為 http://mirexpress.mbc.nctu.edu.tw/index.php
Publication:
1. Wang, W.C., et al., miRExpress: analyzing high-throughput sequencing data for profiling microRNA expression. BMC Bioinformatics, 2009. 10: p. 328
| 圖表六: 高通量序列資料先進行Adaptor序列除去然後與已知miRNA進行序列比對,累計各miRNA所得到的序列數轉換為表現量。 |
● miRNAMap (Nucl. Acids Res., 2006;2008)
miRNAMap: 微小核醣核酸在後生動物基因組中的地圖
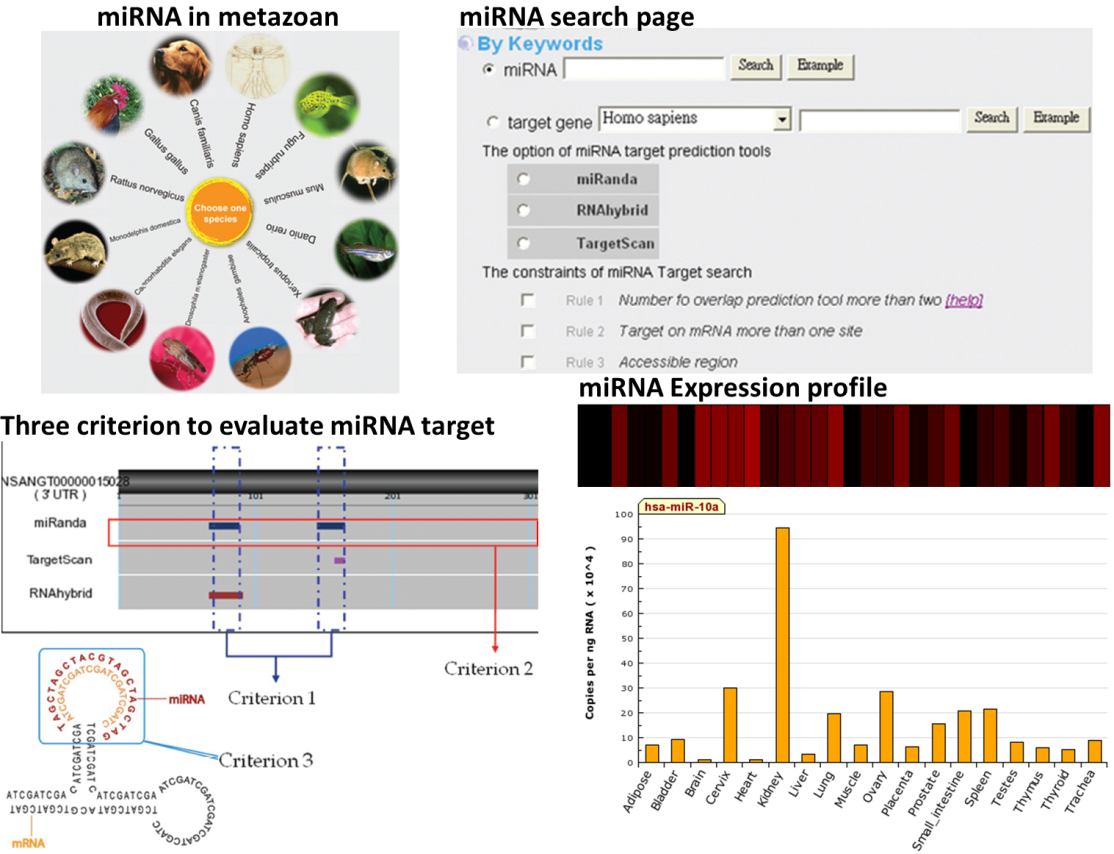
miRNAMap資料庫包含已知與預測的 miRNAs,並利用這些miRNAs預測可能的標靶基因,利用三個準則來提升預測的準確度,包含準則1: 同時被多個工具所預測;準則2: 同一個標靶基因上有無多個target site;準則3: 計算target site的accessibility,即該區域的二級結構是否較不穩定而容易被miRNA target。另外也提供miRNA在不同生物組織中的表現圖譜,此資料可以評估miRNA的組織特異性,如圖表七所示,可以發現此miRNA在腎臟中具高表現量。整體而言,此資料庫因提供了已知的實驗驗證資料和不錯的預測結果,對研究miRNA有極大的幫助。http://miRNAMap.mbc.nctu.edu.tw
Publication:
1. Hsu PW, Huang HD, Hsu SD, Lin LZ, Tsou AP, Tseng CP, Stadler PF, Washietl S, Hofacker IL: miRNAMap: genomic maps of microRNA genes and their target genes in mammalian genomes. Nucleic Acids Res 2006, 34(Database issue):D135-139.
2. Hsu SD, Chu CH, Tsou AP, Chen SJ, Chen HC, Hsu PW, Wong YH, Chen YH, Chen GH, Huang HD: miRNAMap 2.0: genomic maps of microRNAs in metazoan genomes. Nucleic Acids Res 2008, 36(Database issue):D165-169.
圖表七: miRNAMap主要特色。 |
● RNALogo (Nucl. Acids Res., 2008)
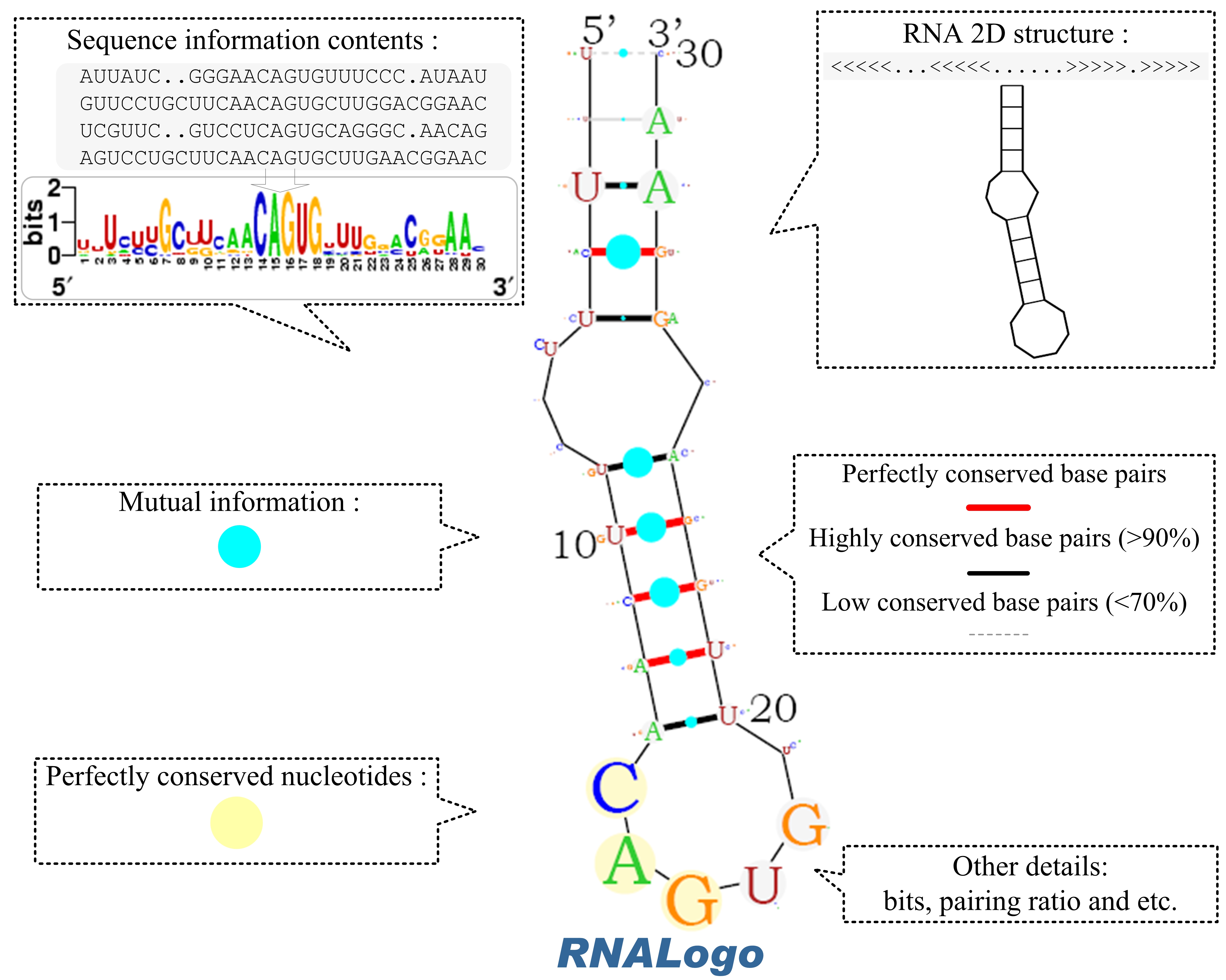
RNALogo是一個呈現RNA二級結構與保留序列之繪圖工具,其圖形呈現方式可有助於使用者了解序列保留性、二級結構保留性等相關資訊於一個RNALogo圖形之中,透過容易使用的網頁介面(http://rnalogo.mbc.nctu.edu.tw/),使用者可以輸入或上傳RNA序列及共同之二級結構,即可繪製其RNALogo圖形(如圖表八所示)。
Publication:
1. T.H. Chang, J.T. Horng, and H.D. Huang. (2008) "RNALogo: a new approach to display structural RNA alignment", Nucleic Acids Research, v.36 W91-6..
圖表八: RNALogo圖示。 |
● ViTa (Nucl. Acids Rea., 2007)
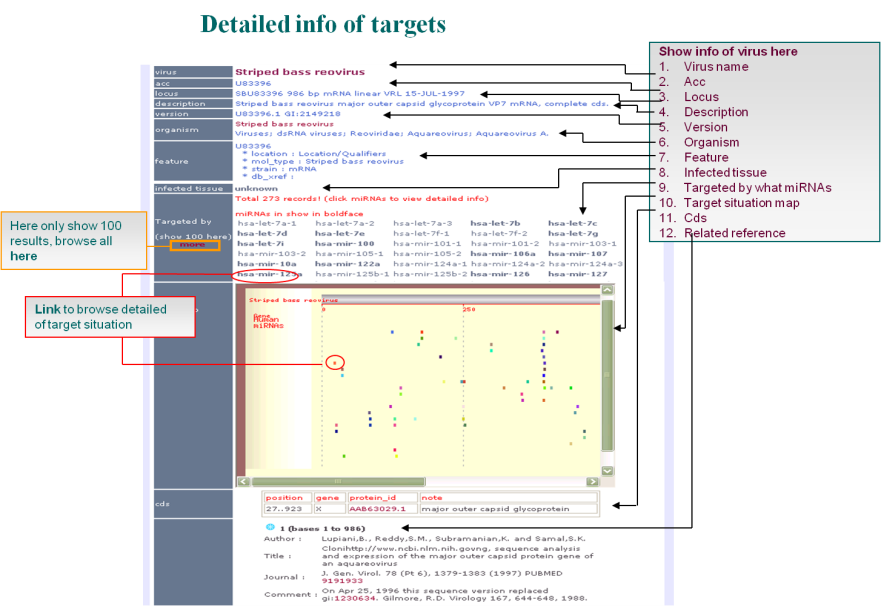
ViTa資料庫是蒐集來自miRBase、ICTV、VirGne、VBRC等資料庫和部份文獻的病毒資料,包括了已知的病毒的miRNAs和利用miRanda和TargetScan工具所預測之宿主的miRNA對於病毒基因體可能的標靶基因(如圖表九)。此外ViTa還提供詳細的註解(如圖表十),包括人類miRNA於不同組織所表現的情況、病毒可能感染宿主的組織等。ViTa網址為 http://ViTa.mbc.nctu.edu.tw
Publication:
1. Hsu PW, Lin LZ, Hsu SD, Hsu JB, Huang HD (2007). ViTa: prediction of host microRNAs targets on viruses. Nucleic Acids Res. 2007 Jan;35(Database issue):D381-5
圖表九: ViTa可提供的所有功能。 |
圖表十: 詳細註解宿主之miRNA於不同病毒基因體的資訊。 |