介紹癌症基因體變異視覺化整合分析工具CoMutPlotter
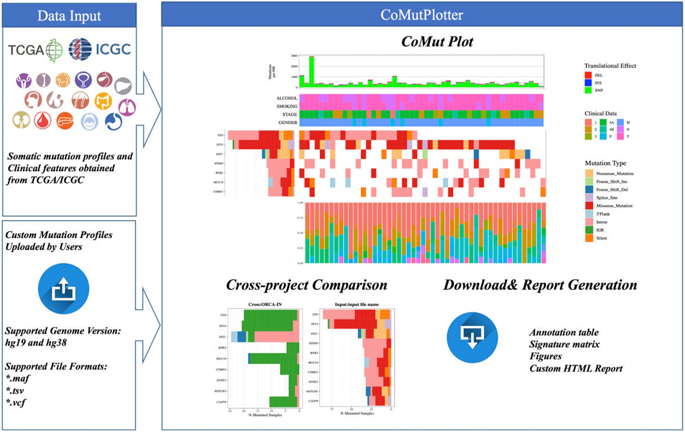
CoMut plot是廣泛應用於癌症基因體研究,利用條狀圖將群體間突變頻率最高的基因由高到低排列出來,同時也可以清楚找出基因體突變最多的個體。此外,也用熱點圖來呈現個體的每個特定基因上突變的程度與變異的種類。最後利用程式語言將這些圖縫合在一起,僅用一張圖就能呈現研究群體中個體間的基因體變異圖譜。看似簡單,但需要經過複雜的檔案格式轉換、變體位點註釋、顯著變異基因預測、變異種類統計分析、突變特徵分析等繁鎖的分析過程。雖然目前已有少數軟體可以用來進行CoMut Plot的製作,但存在著幾個缺點:1.不支援主流的檔案格式(如:VCF格式) 2.缺乏預測顯著變異基因與突變特徵的功能 3.缺乏跨癌症群體比較功能。
有鑑於此,本團隊長庚大學黃柏榕老師實驗室開發了一個網頁工具: CoMutPlotter (http://tardis.cgu.edu.tw/comutplotter/)。使用者不需生物資訊背景就能夠自行操作,上傳研究群體的癌症基因體突變資料,進行全自動分析與產生CoMut plot圖表。CoMutPlotter支援多種基因體變異資料格式(TSV, MAF,VCF),變體位點經過基因功能註解、癌症驅動基因找尋和突變特徵辨認等分析流程,最後將所有結果整合繪製成CoMut plot。除此之外,使用者將自己的研究資料與現有癌症基因體資料庫(TCGA/ICGC)的資料進行比較分析並合併繪圖。使用者能輕易的使用CoMutPlotter比較不同國家的癌症資料差異性。最重要的是,所有比較分析圖表都能讓使用者自由下載。
此工具已於2019年中發表於BMC Medical Genomics期刊。