GEMSiRV: 全基因規模之代謝網路建構、模擬與視覺化平台
代謝網絡模型有助於瞭解生物體的代謝能力與生物特性,但代謝網絡的建構需要同時運用大量的生化、基因體知識並結合數學模型來進行模擬運算,經實驗驗證後,再反覆模擬運算與驗證;然而,目前適合用來建構代謝網絡的相關軟體與資料庫極少,因此TBI核心國衛院群健所熊昭所長、陳豐奇副研究員以及廖玉潔助研究員的研究團隊開發了一個軟體平台,針對全基因體的代謝網絡模型進行模擬、重建與及圖形化介面的呈現 (GEMSiRV: A software platform for GEnome-scale Metabolic model Simulation, Reconstruction and Visualization, http://sb.nhri.org.tw/GEMSiRV)。
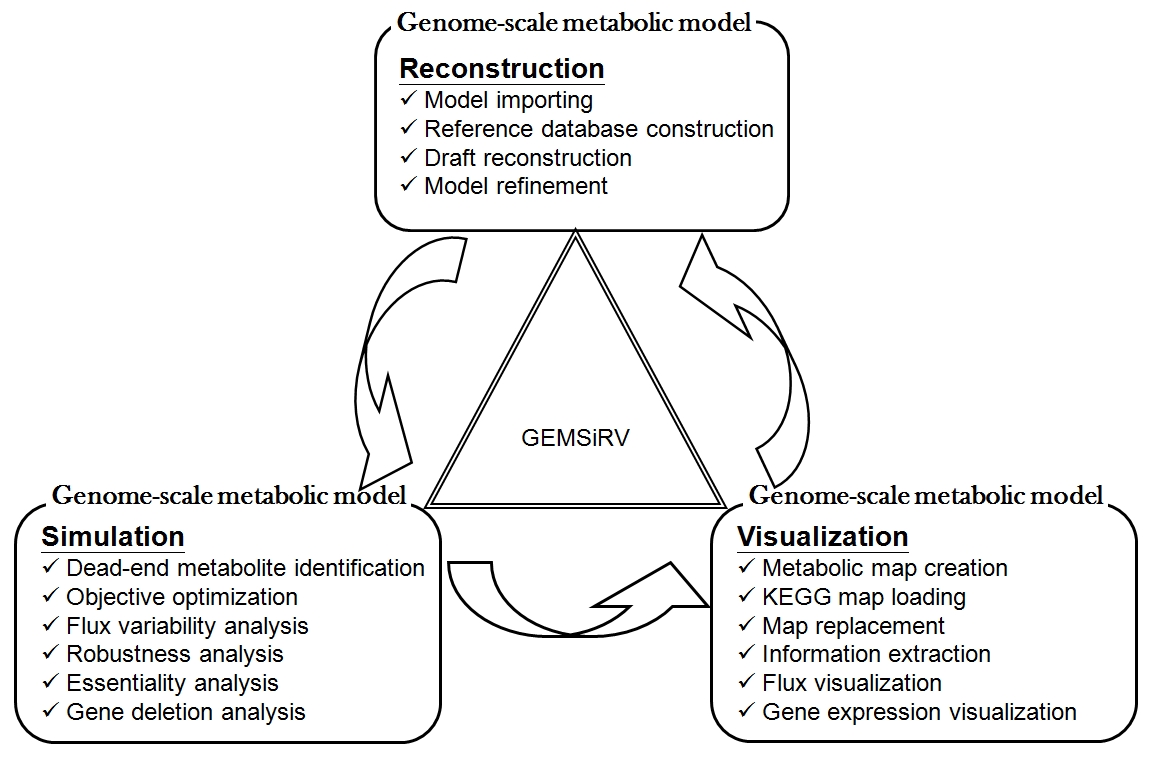
此軟體包含了三個主要的功能架構,分別是網絡重建、模型模擬與圖形化呈現 (圖一)。
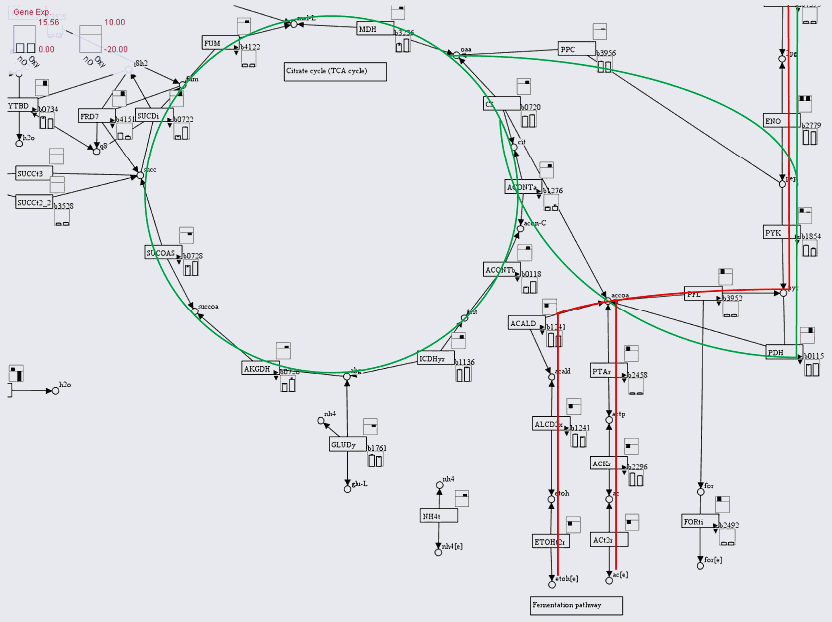
GEMSiRV網站(http://sb.nhri.org.tw/GEMSiRV)上同時提供了代謝網絡建構所需的資料,包括生化反應參考資料庫 (Reference databases) 、過去已經發表的網絡模型 (Metabolic models) 以及使用者自訂的代謝網絡圖 (Metabolic maps),藉由這些資訊的提供,使用者可直接載入GEMSiRV軟體,立即開始進行代謝網絡重建;同時也能載入基因表現量來增加暸解代謝網絡(圖二)。另外,透過比對相近同源基因(orthologous genes),使用者也可利用研究團隊所提供的這些已建構完成的代謝網絡資料,來加速使用者重建新物種的代謝網絡。
圖二 GEMSiRV可載入多種不同資訊以增加使用者對代謝網路的瞭解,除了可以呈現代謝網路的反應流通量(右上角),GEMSiRV還可以載入基因的表現量(右下角),因此可以用來觀察基因表現與網路流通量的一致與否。
我們研究團隊相信,藉由一個整合系統的開發,結合網絡重建、模擬與圖形化介面的功能,可以有效地促進全基因體規模之代謝網絡之建立,並幫助呈現系統生物學之概括,以期能推展並應用於解決實際的生物問題。
Publication:
Bioinformatics (2012) doi: 10.1093/bioinformatics/bts267 First published online: May 4, 2012
http://bioinformatics.oxfordjournals.org/content/early/2012/05/04/bioinformatics.bts267.abstract