以深度學習機制解析巨量腸病毒序列資料並建立基因型鑑別機制與網路即時分析平台
腸病毒(Enterovirus)為台灣一年四季都有發生的常見病毒,為一群病毒家族的總稱,特別好發於兒童,早期症狀都十分相似,很多是沒有症狀的感染,或只出現類似一般感冒的輕微症狀,與一般呼吸道病毒相似,不易判讀。然而其中A71與D68型,常常導致神經系統的併發症等重症的發生。腸病毒為線性的RNA病毒,全長約為7,500個鹼基,具有相當高的突變率,並經常發生基因的重組,目前以單一序列區域來判定型別的檢驗方式,往往容易誤判。同時,採樣後之樣品須送檢至專業核心實驗室處理,待數個工作天後才能獲知型別,往往會讓需要注意的重症患者失去治療的黃金期。
當今由於新世代基因體定序技術的飛躍進步,新一代的單分子定序儀如Oxford Nanopore,體積小且單價低,準確度日益提昇,已可在第一線現場進行定序,同時只需微量DNA樣本與簡單的前處理,便能進行待測基因體群的定序,讓即時的採樣定序已漸漸變為可能,然而,目前尚無線上即時腸病毒全基因體解析與定型平台,可以用來解析定序出來的大量資料,並進一步瞭解樣本中的生態組成(ecosystem)。
由E2生技醫藥生物資訊核心設施熊昭特聘研究員、林介華博士,及中央研究院資訊科學所林仲彥副研究員、陳淑華博士、徐哲仁先生與呂怡萱小姐,所組成的跨領域研究團隊,基於先前在腸病毒多年的研究基礎與腸病毒序列分類資料庫的建置經驗(BMC Genomics, 2015[1]),透過所擁有最為完整且經過校正的腸病毒序列及血清型分型(308型)資料,以深度學習模型來發展更為精準的預測架構,跳脫原先單以病毒外鞘VP1抗體來決定的血清型分型,所遇到的困難與其所導致的可能繆誤。
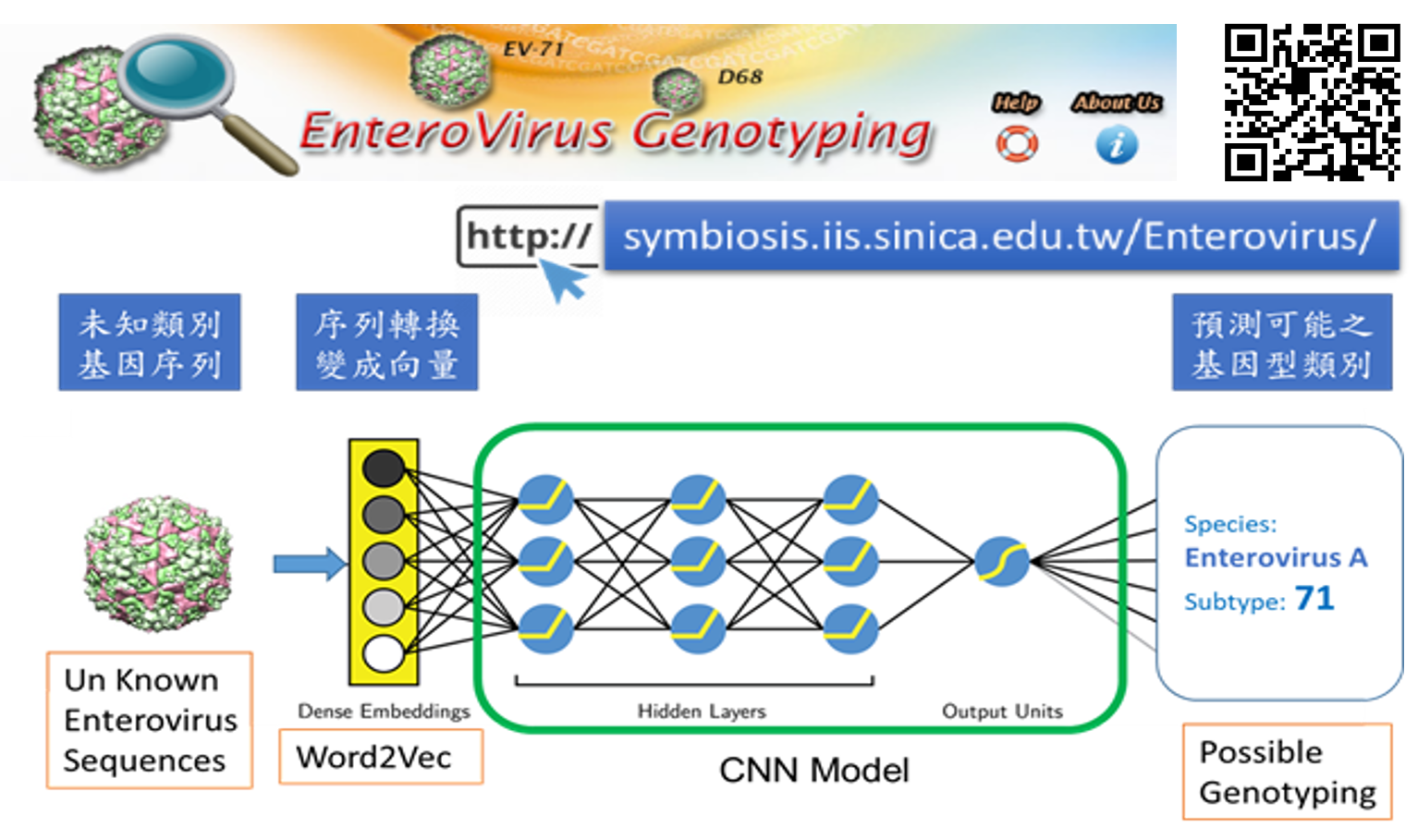
在這個研究中,首先以word2vec藉由3-gram的編碼方式,對腸病毒基因體序列進行高維度向量的轉換,再輸入到所建置的人工智慧模型(EV-CNN)中進行訓練,將所得之深度學習模型,結合網站技術,建構出一個能即時判別腸病毒基因型的線上平台。整體分析的流程,首先透過演算法,先將非腸病毒的序列排除,再依深度學習模型,對序列進行歸類(圖一)。目前的實驗結果顯示,本研究所建置的深度學習模型,如以整體預測率來說,準確度已超過八成,然而針對腸病毒重症品系(如EV-71與D68)來探討,我們的模型的預測準確度可達96%及91%,召回率(Recall rate)分別亦達98%及99%。效能遠遠超過先前僅以VP1來進行序列比對,及利用少數特定資料集所建置的Profile Hidden Markov Models (Profile HMM)模型分類方法,與隨機森林機器學習方法 (與深度學習相同之資料集,90% and 86%)的準確度。以2016-2018台灣疾管局所釋出的腸病毒近六百條的序列為例,利用此一模型平台,可以在數分鐘內完成分類,其預測結果完全與實驗室的分析結果100%一致符合。
透過研究團隊所收集與重新整理的高品質腸病毒序列,及其血清型/基因型等分型資料,利用深度學習的方法,可以建立高準確度的預測模型,並整合網路技術建構線上分析平台,此一線上腸病毒基因型分型平台,將能結合第三代小型低價定序儀(如Nanopore),應用於在腸病毒感染初期(約三到五天內)的檢測上,提供第一線臨床檢體的基因體快速分析流程,即時找出與重症相關的病毒品系,應能及早發現所感染的病毒類型,如屬高致病品系,便能提早準備,防止重症的發生,減少寶貴生命受損的機率,同時也降低可能的醫療費用,並有效防禦及控制疫情,且減低醫療和社會經濟成本,進而實現預防醫學的目標。本成果可應用的領域業別,將包含第一線診所與醫院,生醫檢驗產業,及病毒學研究團隊與流病監控團隊等。目前研究團隊,也正以整個上呼吸道病毒為標的,試著透過類似的策略,來解析呼吸道感染背後所隱含的病原生態組成。此外,本研究成果也榮獲科技部2018年未來科技突破獎的肯定,相關成果亦於去年十二月初的2018未來科技展中展出(圖二)。
關於腸病毒智慧分型線上平台,請參考下列網址:
http://symbiosis.iis.sinica.edu.tw/Enterovirus/

圖二、本研究榮獲2018未來科技突破獎,並接受科技部部長頒獎。
參考文獻:
- Lin C. H., Wang Y. B., Chen S. H., Hsiung C. A.*, and Lin C. Y.*"Precise genotyping and recombination detection of Enterovirus" BMC genomics. 2015 16 Suppl 12, S8. Website: http://symbiont.iis.sinica.edu.tw/evidence