miRTarBase 7.0:強化與升級的miRNA-目標基因資料庫
miRTarBase7.0 (http://miRTarBase.mbc.nctu.edu.tw)是生技醫藥生物資訊核心設施(簡稱BP核心)黃憲達教授與團隊所建構實驗驗證的miRNA-目標基因 (miRNA-target interactions, MTIs) 資料庫。任何研究者可透過miRTarBase 7.0查詢到不同實驗方法所驗證的miRNA調控的基因資訊,也可反向搜尋研究者感興趣的基因會受何miRNA調控。miRTarBase首次發表於2011年,之後歷經過三次重大更新 (Nucleic Acids Res. 2011, 2014, 2016, 2018)。
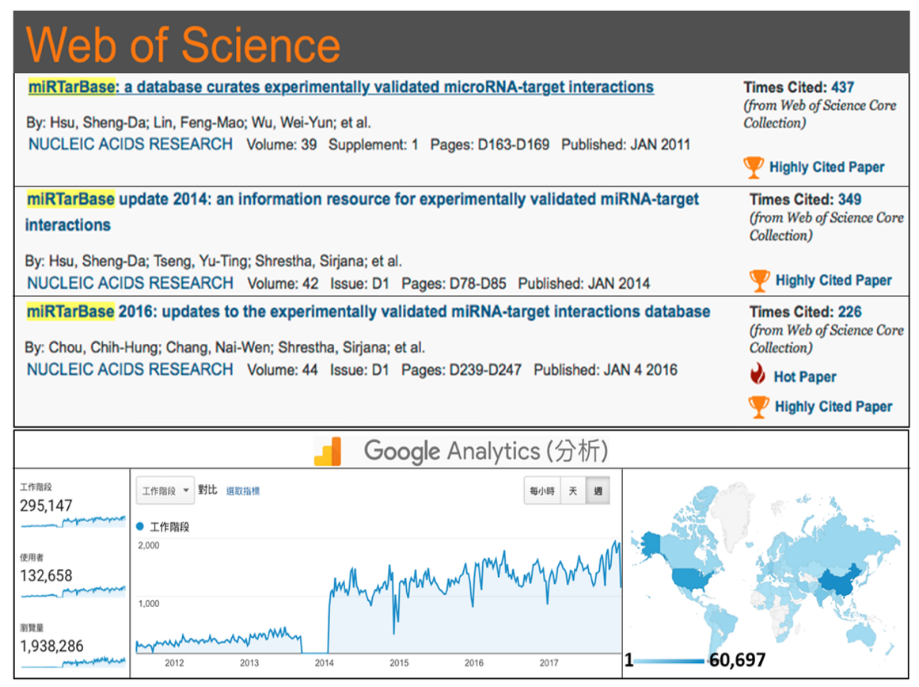
最新版本miRTarBase7.0已於2018年1月於Nucleic Acids Research發表,目前本資料庫包含先前版本總引用次數已超過1,000次,其中2011、2014及2016年版被認定為ESI Highly Cited Paper (圖一),網站瀏覽量超過180萬次。許多國際知名團隊皆使用本資料庫資料進行miRNA相關研究。例如,諾貝爾獎得主Phillip A. Sharp的最新研究中,透過整合miRNA調控網路揭示Super-Enhancer的調控。此外,美國癌症基因體圖譜計畫 (TCGA)團隊,整合miRTarBase資料研究子宮頸癌、胰臟癌、黑色素瘤和甲狀腺癌等。
本資料庫的發表大幅度提升並維繫台灣生物資訊學領域與miRNA研究領域在國際上的能見度。miRTarBase7.0易於使用,生物實驗學家可藉由簡單明確的使用介面,快速的查詢到不同物種中已知miRNA所調控之基因資訊,而生物資訊學家也能取得所有資料庫內容,應用於觀察與重建miRNA基因調控網路,更能藉由miRNA表現趨勢與所控制之目標基因解析miRNA在調控特定生物代謝路徑與不同癌症或複雜疾病之致病機制全貌。
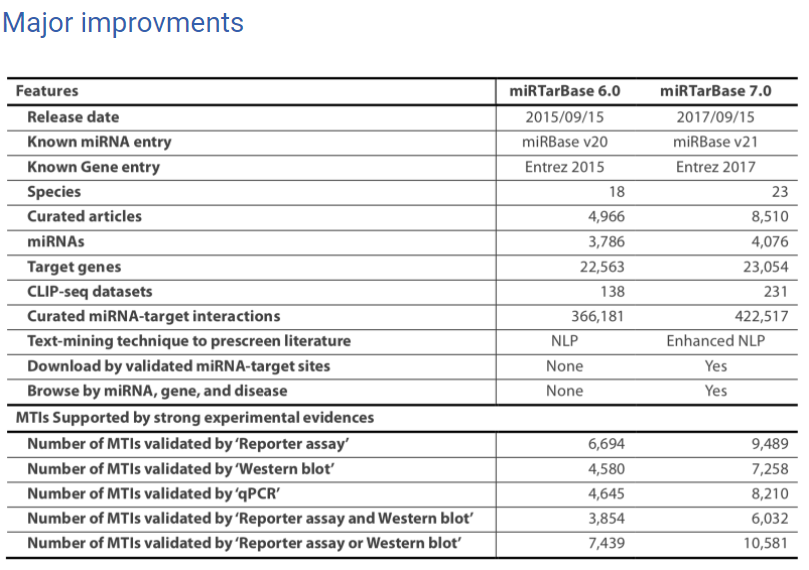
miRTarBase7.0資料庫目前由超過8,500篇發表之SCI學術論文中,共計23個不同物種中總數4,076個miRNAs與23,054個基因中,收錄到422,517筆miRNA與目標基因交互關係 (圖二),並持續整理與收錄。其它新版本所提升的功能包含了強化miRNA-疾病文字雲讓使用者更加迅速辨識miRNA與疾病之關聯、增加CLIP-seq定序資料數量並提供詳細target site資訊、更新TCGA 臨床資料與miRNA與不同癌症之相關性。