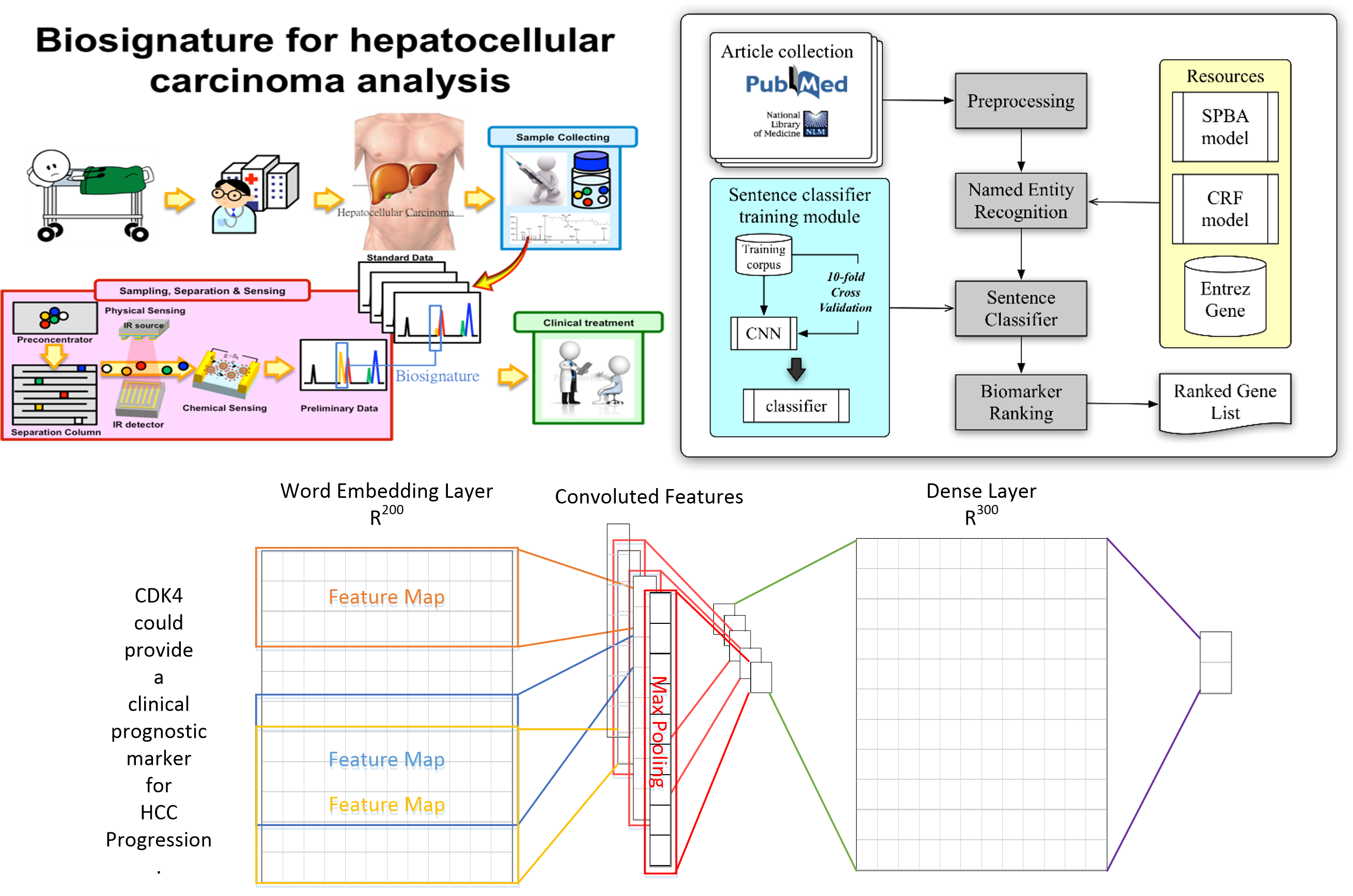
以文獻探勘技術從巨量文獻數據庫中挖掘肝細胞癌的生物標識
PubMed是由美國的National Center for Biotechnology Information所開發,存放最多生醫文獻,也是生醫研究人員在研究過程中最依賴的資料庫。所有珍貴的研究成果都被完整保存,資料庫中每年所收集的文獻數量呈現正成長,根據統計,2017年將會有超過一百二十萬篇研究成果被納入資料庫中,平均每天約有3,300篇。隨著科技的發展,這樣的研究能量將會逐年遞增。研究人員該如何從如此巨量的文獻資料中找到與自己研究相關的文獻,文獻的蒐集及分析是現在正面臨的一大問題。
肝細胞癌(Hepatocellular carcinoma, HCC)是死亡率相當高的癌症之一,其5年生存率為18%,且治療方針需要非常強大的生物標誌(Biosignature)。儘管HCC的相關研究文獻逐年遞增,但許多候選生物標誌都尚未得到臨床驗證。為了縮小候選生物標誌的範圍,以便進一步進行臨床驗證,本研究構建了MarkerHub,從PubMed收集2008~2017的已發表的生醫文獻摘要,以類神經網路演算法篩選出具有HCC候選基因的句子後,利用自然語言處理的命名實體辨識技術進行基因/蛋白質名稱辨識。最後透過全域排序演算法,針對擷取出的各種關鍵因子,如文章的重要性及關鍵詞頻率等,計算候選基因的可信度,替候選生物標識進行排序。再結合蛋白質交互作用資訊,提供HCC候選生物標誌相關的蛋白質網路。此方法將能輔助生醫專家快速掌握HCC的最新成果及全面性的研究發展。本系統已開放 (http:// http://markerhub.iis.sinica.edu.tw) 提供使用。