從多源基因體序列資料中有效地組裝出病毒全基因體工具之開發
Hsin-Hung Lin and Yu-Chieh Liao*, drVM: a new tool for efficient genome assembly of known eukaryotic viruses frometagenomes. GigaScience 6, 1-10 (2017).
現今使用高通量的次世代定序技術來解析樣本中的病原基因體方法已漸趨成熟,因此可以應用來鑑識出樣本中的病原體,然而實際上操作、分析這樣的序列資料是需要先配置特定的分析工具,再經過繁瑣的分析步驟與一連串的資料解析程序才能夠獲得最終想獲得的病原體資訊。有鑒於此,開發一套友善使用者並具有圖形化操作介面的工具是必要的,E2轉譯醫學暨生技研發之生物資訊核心設施熊昭研究員團隊開發本系統drVM可以有效地組裝多源基因體 (metagenomes) 序列資料中的病毒基因體,本程式已包裝成虛擬機器 (virtual machine) 方便使用者直接下載、匯入後使用,另外也提供進階使用者直接使用程式碼和各種通用的映象檔包括Amazon machine image以及docker。
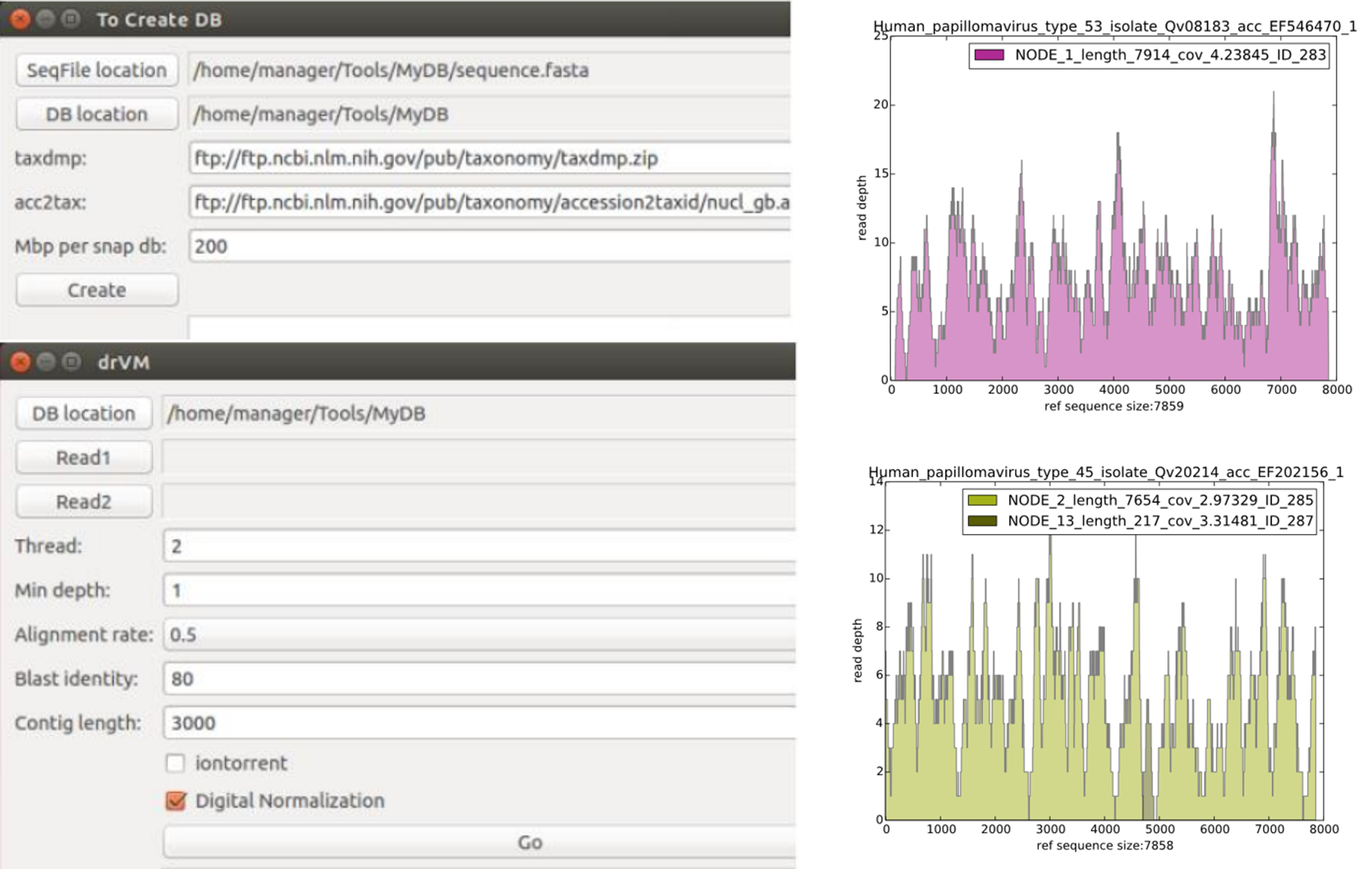
此套自動化分析工具的分析程序,主要提供了病毒基因體資料庫建置的程式CreateDB.py以及病毒序列分析的主程式drVM.py,可以讓使用者自行建構最新的病毒基因體資料庫,並快速地組裝出樣本中所含有的病毒基因體序列,最終產出容易解讀的資料型態,提供病毒序列對應至組裝基因序列上的分布圖譜 (coverage profile),幫助使用者瞭解樣本中該病毒的序列深度以及組裝序列的正確性與完整性。舉例說明,本工具分析人類微生物基因組學計畫 (Human microbiome project, HMP) 中的樣本,可以自動化組裝出兩條完整的不同型別卻共存的人類乳突病毒 (Human papillomavirus, HPV) type 45以及type 53的全基因體病毒序列。
此工具實際應用在分析超過三百組的多源基因體序列,成功地組裝出各式的病毒基因體序列。比較本工具和其他類似工具的結果,本工具在操作上、執行速度以及組裝序列的完整性都具有絕佳的優勢,因此相信可以應用於臨床樣本中新興病毒的鑑定。本自動化分析工具已上線 (https://sourceforge.net/projects/sb2nhri/files/drVM/) 提供免費下載。
