蛋白質結構套裝分析流程
在蛋白質結構研究與分析應用主題方面,我們在這次的結構生物資訊研習會(103年9月9日)提供了系統性的概念與操作學習。主題結合本核心自行研發的生物資訊資料庫與工具,套裝規劃課程內容,以實際蛋白質研究主題引導學員切入學習,自蛋白質基礎結構解析開始,教導學員如何利用蛋白質結構視覺化軟體瞭解蛋白質的結構特性,接著進行蛋白質結構模擬、蛋白質結構預測、蛋白質結構比對與電腦輔助藥物設計(包含分子建構、對接與虛擬篩選)等一系列的研究流程,提供學員系統性的概念與操作學習,使其具備實際應用相關生物資訊工具分析蛋白質相關功能特性與藥物設計基礎的能力。以下是摘要整理本次研習會的內容:
一、蛋白質結構分析與結構視覺化軟體操作
後基因體時代大部分的研究都圍繞在Genomics、Proteomics、Metabolomics、Lipidomics等領域,但結構生物學在每一主題的研究上皆扮演著重要的角色,以電腦模擬輔助及生物資訊的分析,可以輔助研究者全面性的了解其生物分子的結構與功能特性,同時進一步運用這些資訊與研究成果,發展相關藥物設計的研究,由基礎的生物巨分子結構,協助研究者探討生物分子結構與功能的關係,推廣到分子層次及細胞層次藥物醫學的研究應用。
由於基本的蛋白質結構分析是學習結構生物學的基礎,解讀蛋白質資料庫(Protein Data Bank, PDB)是基本不可或缺的內容,學習的項目包括PDB資料庫的統計資料、結構頁面的主要分類內容說明(Summary, 3D View, Sequence, Annotations, Seq. Similarity, 3D Similarity, Biol. & Chem., Methods, Links),同時下載pdb結構檔案,詳細說明其格式與定義。
第二部分是結構視覺化軟體操作練習,Molecular graphics softwares有非常多,像是RasMol, PyMOL, Cn3D, Swiss PDB viewer, MOLMOL, MolScript, Raster3D, Chimera等,結構視覺化的軟體是一種顯現生物巨分子結構的軟體,可轉換相關原子的空間座標,包含蛋白質、DNA、RNA、化學小分子和金屬等皆可藉由視覺化軟體來呈現其結構。利用結構視覺化軟體可以輔助觀察巨分子的結構、作用力、表面特性等,尤其在藥物設計、分子模擬上有很大的應用空間。因此,至少學習1-2種結構視覺化軟體操作與熟練,將會在蛋白質結構分析方面達到事半功倍的效果。

本次課程提供學員學習基本和進階的PyMOL操作技巧,製作高品質的蛋白質3D結構圖形,PyMOL也是發表論文最常使用的蛋白質結構視覺化軟體之一,目前學術上使用免費,但是要用之於發表就需要付費才行。
另外,我們也提供了有關蛋白質結構的資料庫或工具連結,讓大家可以互相參考應用:
|
PDBsum is a pictorial database that provides an at-a-glance overview of the contents of each 3D structure deposited in the Protein Data Bank (PDB). It shows the molecule(s) that make up the structure (ie protein chains, DNA, ligands and metal ions) and schematic diagrams of the interactions between them. |
|
PROSITE: Database of protein domains, families and functional sites PROSITE consists of documentation entries describing protein domains, families and functional sites as well as associated patterns and profiles to identify them. PROSITE is complemented by ProRule, a collection of rules based on profiles and patterns, which increases the discriminatory power of profiles and patterns by providing additional information about functionally and/or structurally critical amino acids. |
|
|
Prediction of transmembrane helices in proteins |
|
|
ProtScale allows you to compute and represent the profile produced by any amino acid scale on a selected protein. An amino acid scale is defined by a numerical value assigned to each type of amino acid. The most frequently used scales are the hydrophobicity or hydrophilicity scales and the secondary structure conformational parameters scales, but many other scales exist which are based on different chemical and physical properties of the amino acids. This program provides 57 predefined scales entered from the literature. |
|
|
InterPro is an integrated database of predictive protein signatures used for the classification and automatic annotation of proteins and genomes. InterPro classifies sequences at superfamily,
family and subfamily levels, predicting the occurrence of functional domains,
repeats and important sites. InterPro adds in-depth
annotation, including GO terms, to the protein signatures. |
|
Pfam 26.0 The Pfam database is a large collection of protein families, each represented by multiple sequence alignments and hidden Markov models (HMMs). |
|
|
The free, collaborative 3D-encyclopedia of proteins & other molecules. |
二、 蛋白質結構模擬與預測
了解蛋白質的基礎結構特性與學習基本的結構視覺化軟體操作之後,便可以針對自己手上的蛋白質進行初步的模擬與預測。我們用的蛋白質結構預測軟體是本核心成員(交通大學)所開發的 (PS)2: Protein Structure Prediction Server,網址在http://ps2.life.nctu.edu.tw/ ,(PS)2可以偵測較遠的同源蛋白質,並且使用多重的模版預測蛋白質結構。(PS)2的使用方式非常簡單,首先是貼上你的蛋白質序列,樣板可以選擇Automatic或是直接指定要用的Template,然後送出。如下圖:

本例是直接指定以3TE4_A為Template,得到下列結果:
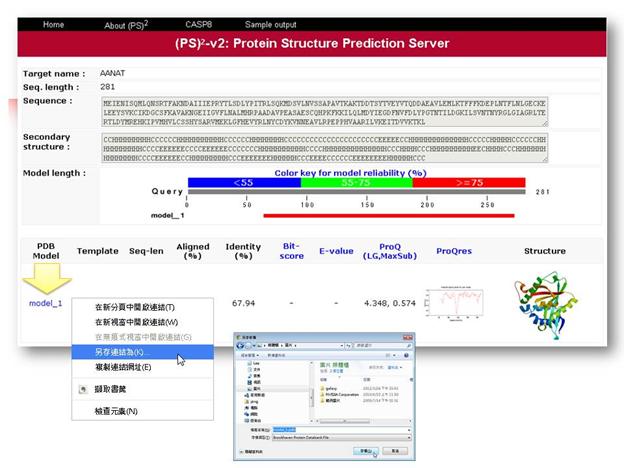
將模擬的結構model_1.pdb儲存,並以PyMOL開啟:
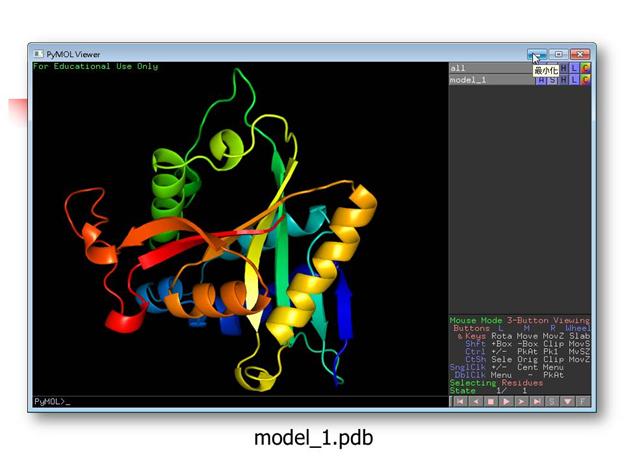
模擬出來的結構記得可到PDBsum, http://www.ebi.ac.uk/pdbsum/,利用PROCHECK檢視其Ramachandran plot:
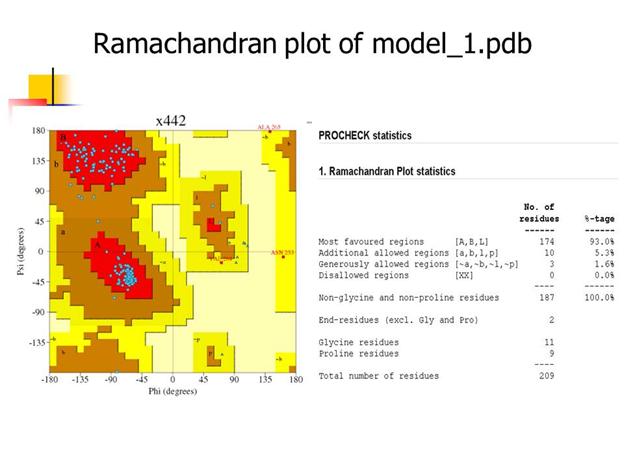
三、 利用結構比對方法搜尋相近的蛋白質
得到模擬的蛋白質結構後,可以利用結構比對方法搜尋相近的蛋白質,了解相關蛋白質的功能特性。使用的蛋白質結構比對軟體是本核心成員(清華大學)所開發的iSARST,網址在http://sarst.life.nthu.edu.tw/iSARST/,iSARST在多工而平行化的作業環境下整合了數種結構比對工具及兩個高效率的搜尋方法:適合尋找一般類似物的 SARST 及專門搜尋環形序列重組類似物的CPSARST。iSARST允許使用者一次輸入多筆蛋白質結構。資料輸入後,伺服器會先以SARST/ CPSARST搜尋結構資料庫,再用高準確率的結構比對軟體重新計算所得蛋白質與查詢蛋白質間的結構相似度並按相似度大小列出清單,以求快速回報資料並確保所得資訊的高準確度。iSARST並提供互動式結構檢視介面以利使用者做進一步分析、研究。預期該伺服器可為後基因體時代的結構生物學家們提供方便的工作平台。iSARST的輸入介面如下圖:
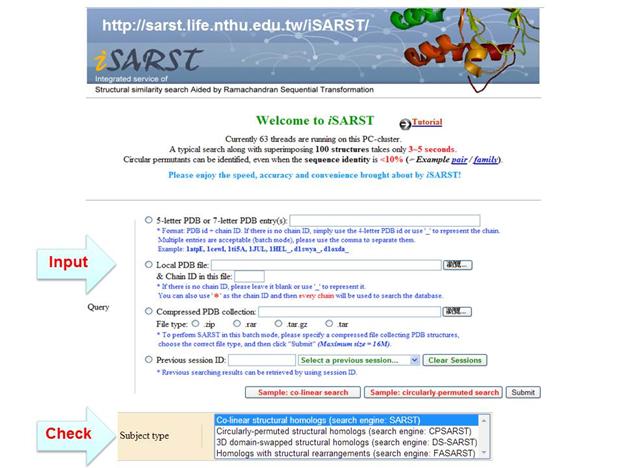
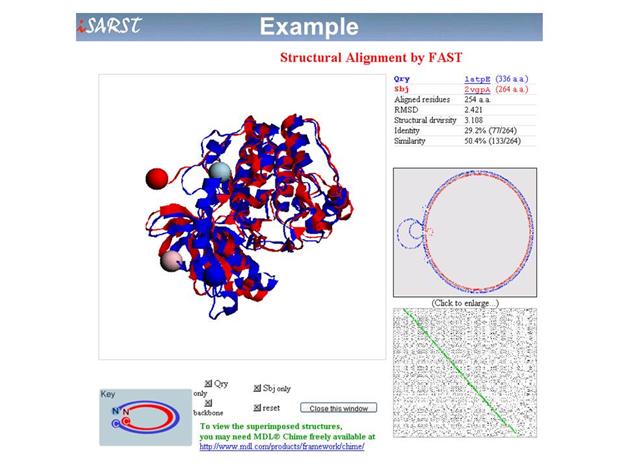
輸出的互動式結構檢視頁面如下:
四、 分子對接 (Molecular Docking)
在生物體中,要進行生化、催化或代謝反應時須要酵素(enzyme)和其接受體(substrate, ligand)結合後進行反應,其範圍包含甚廣。例如訊號傳遞(signal transduction)、荷爾蒙作用、脂肪酸的運送等。因為不同分子之間的彼此交互作用,例如蛋白質-蛋白質、蛋白質-核酸、蛋白質-脂肪酸等,而引發生許許多多的特定生化功能。當我們利用電腦來模擬各種結合的情形時,我們稱這一種技術為嵌合(docking)。嵌合方法在結構生物上的應用就是在藥物設計方面,找出可能的活化區(active site)、找出前驅物(lead compound)、突變(mutation)的影響等。利用此一方法可以有效率的減少在實驗上、設計上所須要花費的時間。
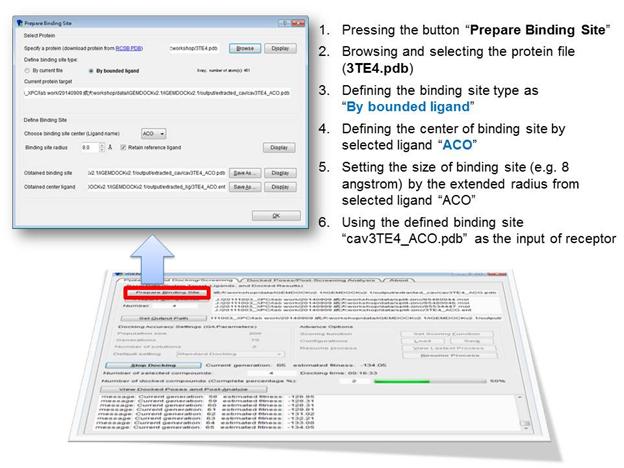
我們所使用的分子對接軟體是本核心成員(交通大學)所開發的iGEMDOCK,用於ligand與active site分析,網址在http://gemdock.life.nctu.edu.tw/dock/igemdock.php。iGEMDOCK的使用以圖形視窗介面呈現,首先是Prepare Binding Site,選擇目標的蛋白質,如下圖:
接著是Prepare Compounds指定Ligand file,iGEMDOCK可以接受的檔案格式為MDL MOL、SYBYL MOL2和PDB format,建立Ligand file時建議可以參考ZINC資料庫(http://zinc.docking.org/)的建構化學式和格式轉換功能,例如像是PDB資料庫所提供Ligand的SMILES格式即可利用ZINC來轉換輸出成各種檔案格式。
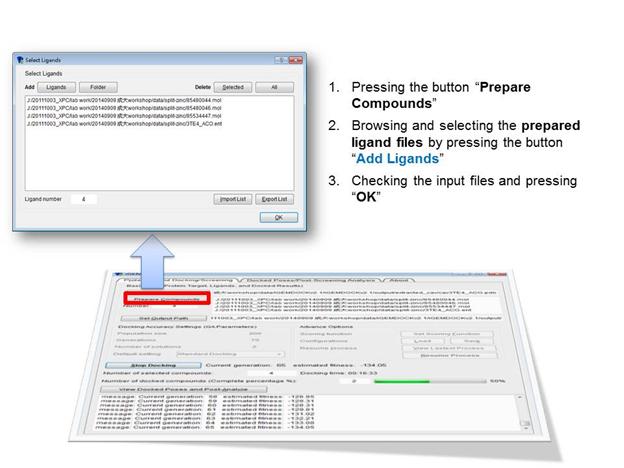
接下來是Setup output path,設定Docking結果輸出的路徑,建議每次重新設定,以免先前資料結果被覆蓋消失。
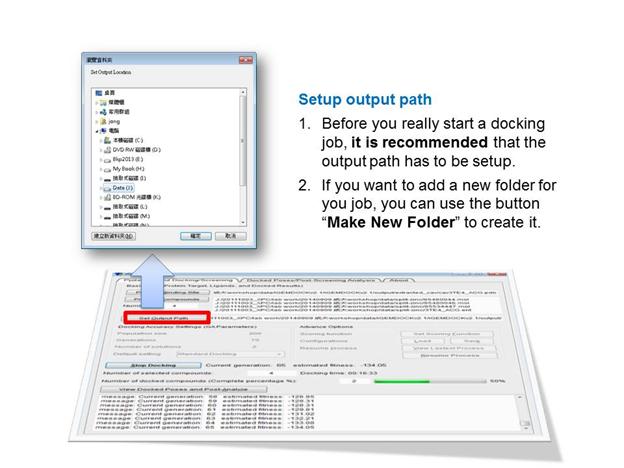
接下來是Setup GA Parameters,設定Population size、Generations和Number of solutions,此處請參考各自電腦的配備,相關設定會影響結果輸出的時間。
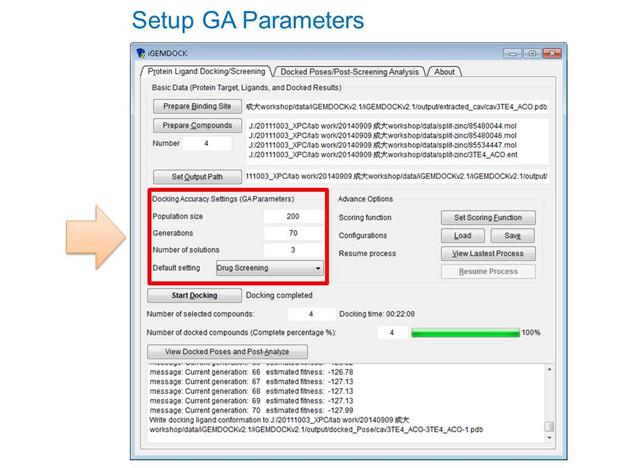
設定完成後,即可進行Start Docking,Docking完成的時間會顯示在右邊,下方有View Docked Poses and Post-Analyze選項可供檢視Docking結果和後續分析。
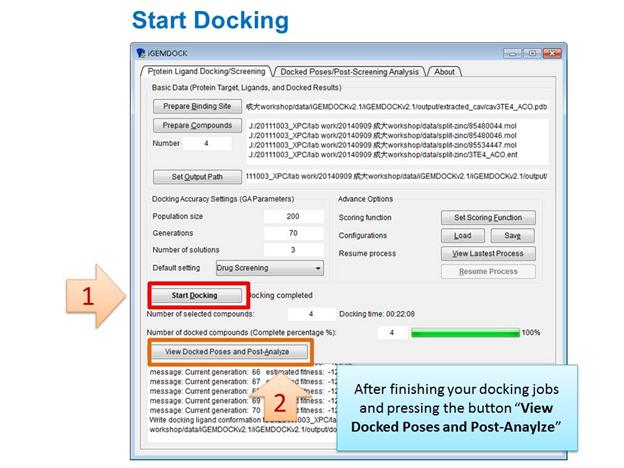
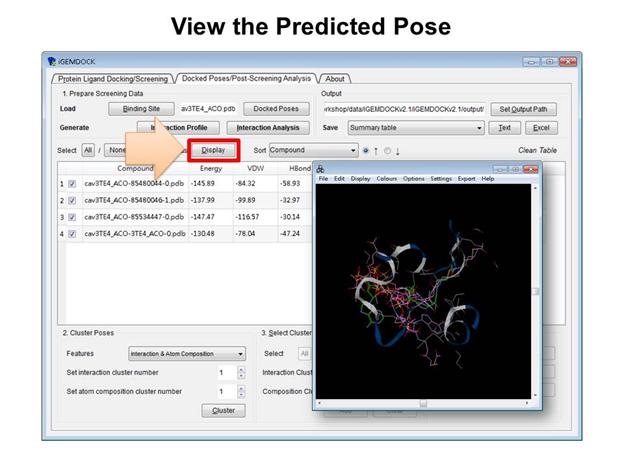
下圖是檢視Docking的結果,勾選其中Docking的結果,再點選Display選項,iGEMDOCK會直接以Rasmol軟體顯示對應的立體結構,相當方便。
最後,我們利用下圖做一個總結,要善用結構生物資訊分析工具之前,必須先對蛋白質基礎結構特性有充分的了解, 同時熟悉蛋白質結構視覺化軟體操作,再針對感興趣的蛋白質目標進行結構模擬與預測、結構比對與分子對接等一系列的研究流程,俾有利於探討後續蛋白質的功能特性與藥物設計相關主題。當然上述的流程較為制式化,但若配合相關位置突變或是Circular permutation (CP) site的設計,也會增加整個結構生物資訊分析流程的多樣性與功能性,這部分則需要與使用者共同討論與發展,大家若有需求的話歡迎與我們聯繫。
蛋白質結構整合分析客製化服務
我們有完善蛋白質結構整合分析客製化服務平台,包含一系列蛋白質結構研究分析,自蛋白質基礎結構解析開始,從如何利用蛋白質結構視覺化軟體瞭解蛋白質的結構特性,接著進行蛋白質結構模擬、蛋白質結構預測、蛋白質結構比對與電腦輔助藥物設計 (包含分子建構、對接與虛擬篩選) 等一系列的研究流程,到NMR或X-ray結構分析等研究支援,同時整合我們已發展之蛋白質結構分析比對工具及分子設計軟體,提供使用者研究蛋白質結構與功能的最佳解決方案。尤其是在目前因大量基因序列被解碼,但對基因轉譯後蛋白質的真正生物功能仍無完整了解的情況下,此平台可因應與協助研究者對未知蛋白質結構與功能的研究需求。
另外,在蛋白質-化合物與藥物設計客製化服務方面,利用我們自行研發的的iGEMDOCK及SiMMap以及統計分析專長,分析蛋白質-化合物關係,可運用於藥物開發,如尋找可能的Multi-target的多目標藥物及舊藥新用。本服務將發展為蛋白質-化合物整合型分析服務,可針對特定疾病提供相關蛋白、生化途徑與化合物(藥物、天然物);針對特定蛋白提供相關疾病與化合物(藥物、天然物);針對特定化合物提供潛在標靶蛋白及其所參與之生化途徑與疾病。
TBI 核心設施提供蛋白質結構分析服務,內容包括提供線上分析工具與客製化資料分析服務,TBI核心設施提供多種蛋白質結構分析工具,為免費的網路服務,研究人員可直接上網使用,若有研究成果發表,可將分析工具列於材料與方法,並於參考文獻引述該項分析工具的論文或網址;如需客製化結構資料分析服務(如modeling、molecular docking等),只要填寫申請表Email至服務信箱即可,我們會與您聯絡安排需求訪談,如有任何問題,歡迎隨時利用服務專線洽詢:
Email: service@tbi.org.tw
客製化服務申請表(下載)