蛋白質結構分析服務
TBI 核心設施提供蛋白質結構分析服務,內容包括提供線上分析工具,以及客製化資料分析服務,TBI核心設施提供多種蛋白質結構分析工具,為免費的網路服務,研究人員可直接上網使用,若有研究成果發表,可將分析工具列於材料與方法,並於參考文獻引述該項分析工具的論文或網址;如需客製化資料分析服務,只要填寫申請表email至服務信箱即可,我們會與您聯絡安排需求訪談,如有任何問題,歡迎隨時利用服務專線洽詢:
服務專線: 037-206166 x33621
Email: service@tbi.org.tw
客製化服務申請表(下載
Word DOC
PDF )
一、蛋白質結構線上分析工具:
a.蛋白質結構分析工具
1) CELLO:蛋白質細胞定位預測工具。http://cello.life.nctu.edu.tw/
2) (PS)2–V2,蛋白質結構預測:可以偵測較遠的同源蛋白質,並且使用多重的模版預測結
構。http://ps2.life.nctu.edu.tw/
3) KinasePhos,蛋白質磷酸化預測資料庫http://kinasephos.mbc.nctu.edu.tw/
4) dbPTM,蛋白質後轉譯修飾資料庫 http://dbPTM.mbc.nctu.edu.tw/
5) 3D-BLAST (快速蛋白質搜尋工具) (http://3d-blast.life.nctu.edu.tw/) 和FastSCOP (蛋白質功能分類)
(http://fastscop.life.nctu.edu.tw/):
6) pKNOT (環結拓樸特質蛋白質分析工具) (http://pknot.life.nctu.edu.tw/)
7) CPDB : http://sarst.life.nthu.edu.tw/cpdb/ 蛋白質的環形序列資料庫
8) 蛋白質結構快速搜尋比對工具iSARST : http://sarst.life.nthu.edu.tw/iSARST/
9) CPSARST : http://sarst.life.nthu.edu.tw/cpsarst 高效能蛋白質環形序列重組搜尋比對工具
10) Tm Predictor : http://tm.life.nthu.edu.tw/ 預測當ㄧ半的雙股 DNA 解離開來時的溫度
11) GPDB : http://gpdb.life.nthu.edu.tw/ 基因體分析比較工具與資料庫
12) 2D virtual gel : http://gpdb.life.nthu.edu.tw/V2D/ 虛擬二維電泳資料庫
b.電腦輔助藥物設計分析工具務
1) GEMDOCK 分子對接軟體,用於ligand與active site分析,提供使用者下載使用http://gemdock.life.nctu.edu.tw/dock/
2) SiMMap 藥物設計工具 (http://simmap.life.nctu.edu.tw/):提供蛋白質與嵌合化合物在結合環境中交互作用的關係,
可找出特定蛋白質中化合物結合位之熱區(hot spot)。
二、蛋白質-化合物與藥物設計整合型分析客製化服務
利用我們自行研發的的iGEMDOCK及SiMMap以及統計分析專長,分析蛋白質-化合物關係,運用於藥物開發,如
尋找可能的Multi-target的多目標藥物及舊藥新用。本服務將發展為蛋白質-化合物整合型分析服務,可針對特定疾病
提供相關蛋白、生化途徑與化合物(藥物、天然物);針對特定蛋白提供相關疾病與化合物(藥物、天然物);針對特
定化合物提供潛在標靶蛋白及其所參與之生化途徑與疾病。
三、蛋白質結構整合分析客製化服務
包含一系列蛋白質結構研究分析,從序列比對、註解、結構預測與模擬,到NMR或X-ray結構分析等研究支援,
提供使用者研究蛋白質結構的最佳解決方案。同時,針對微生物結構基因體資料庫(Structural Genomics Databases),
我們亦可協助整合建立基因體及結構分析平台,相關內容請參考網址: http://bioinfo.life.nthu.edu.tw/microbial/。
蛋白質結構分析工具介紹
CPred分析工具
http://sarst.life.nthu.edu.tw/CPred/
蛋白質的環形序列重組 (Circular Permutation, 簡稱CP)可看作是其原本的N與C端被相接,然後在另一處產生開口,顯示在演化時,結構同源的蛋白質以不同位置作為終端點(termini)。由於CP通常在結構與活性上具有較佳的穩定性(stability)、活性(activities)或是功能的多樣性(functional diversities),以蛋白質工程的方法製造出CP,已被應用於許多相關領域。考量並非每一個位置都適合製造出具有活性的CP,且用分子選殖(molecular cloning)的方式製造CP的成本很高,預測CP 位置遂成為重要的工作。我們因此發展出一預測CP位置的分析工具CPred (Circular Permutation site Predictor),以促進應用CP進行研究的效率。
一、CP預測方法
CPred 利用四種 machine learning 方法,包括 artificial neural network (ANN)、support vector machine (SVM)、random forest 以及hierarchical feature integration來預測CP。CPred會計算蛋白質中每一個胺基酸形成CP的機率值,因此機率值愈高者表示該位置形成具有活性的CP的機率愈高。
二、CPred使用步驟
Step 1 輸入欲查詢之蛋白質
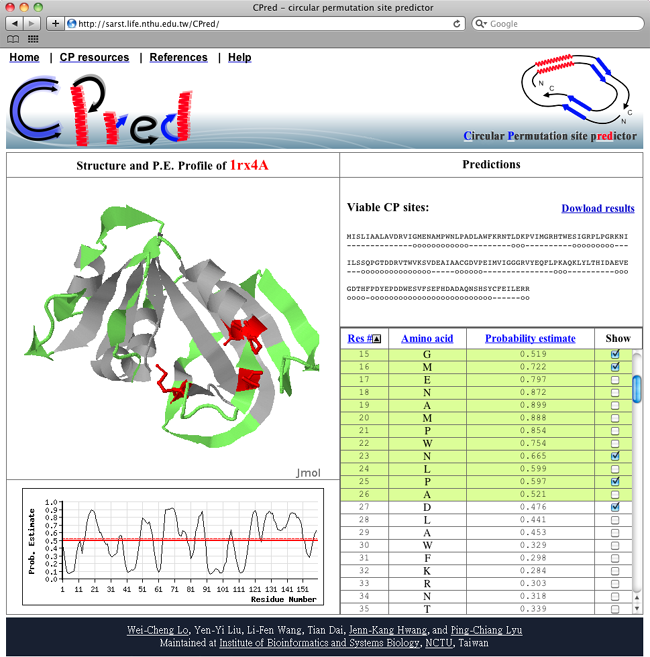
您可輸入PDB/SCOP蛋白質識別碼(ID)或上傳一PDB file,並選擇是否需要按照機率排序,即可展開預測。
Step 2 由CPred進行預測
Step 3 CP預測結果:
CPred提供互動式的 Jmol applet 來展示預測出來的 CP sites,預設是以綠色表示。下圖左下方的曲線圖顯示蛋白質序列中可能形成 CP的機率,高於紅色實線(估計值 >0.50)者表示預測可形成CP,而紅色虛線(估計值 >0.52)者表示可能形成有功能的CP。
三、參考文獻:
Lo WC, Wang LF, Liu YY, Dai T, Hwang JK, Lyu PC: CPred: a web server for predicting viable circular permutations in proteins. Nucleic acids research 2012, 40: W232–237. [Link]
Lo WC, Lee CC, Lee CY, Lyu PC: CPDB: a database of circular permutation in proteins. Nucleic Acids Research 2009, 37:D328-332. [Abstract] [PDF]
Lo WC, Lyu PC: CPSARST: an efficient circular permutation search tool applied to the detection of novel protein structural relationships. Genome Biology 2008, 9:R11. [Abstract] [PDF]
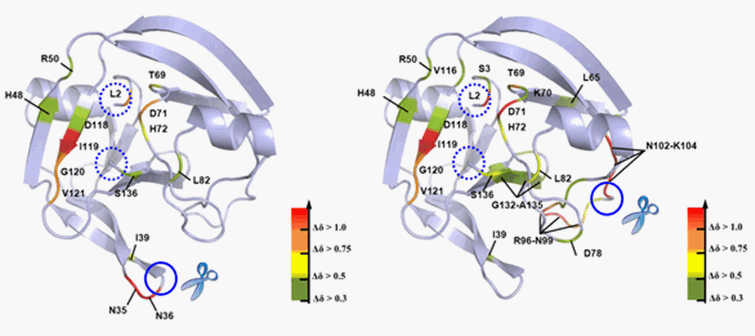
CPred應用實例參考
RESEARCH ARTICLE
Circular Permutation Prediction Reveals a Viable Backbone Disconnection for Split Proteins: An Approach in Identifying a New Functional Split Intein.
Lee YT, Su TH, Lo WC, Lyu PC, Sue SC (2012) PLoS ONE 7(8): e43820. doi:10.1371/journal.pone.0043820 [Abstract] [PDF]
Dotted circles indicate the locations of the N- and C-terminal ends, and closed circles indicate the introduced CP sites.